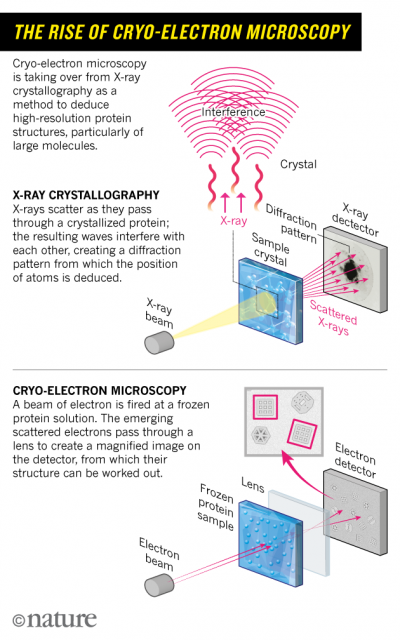
La criomicroscopia electrónica ha permitido ver ciertas biomoléculas con una resolución atómica. Por su desarrollo han recibido el Premio Nobel de Química Jacques Dubochet (Univ. Lausanne, Suiza), Joachim Frank (Univ. Columbia, New York, EE.UU.) y Richard Henderson (Lab. Biología Molecular MRC, Cambridge, Reino Unido). Yo llevo varios años recibiendo en mi despacho la revista Nature Methods y uno de los temas que más me asombra son las nuevas tecnologías en microscopia que permiten inferir la estructura de una proteína átomo a átomo.
El microscopio electrónico recibió el Premio Nobel de Física de 1986 (Ernst Ruska en solitario). Su gran problema era que no podía observar biomoléculas (moléculas de interés bioquímico sin destruirlas). La solución parecía sencilla de concebir, usar muestras formadas por una sola biomolécula enfriada a muy baja temperatura, pero ponerla en práctica ha constado décadas de investigación. La clave era preservar intacta la biomolécula en su estado hidratado y usar una irradiación electrónica que no fuera destructiva. Muchos fueron los pasos que dieron otros investigadores y que allanaron el camino a los galardonados.
Más información en el resumen, el anuncio del galardón, la nota de prensa, la información divulgativa y la información científica.
[PS] Gracias a César Tomé por aclararme que «microscopia crioelectrónica» es un término sin sentido en español, siendo lo correcto «criomicroscopia electrónica». [/PS]
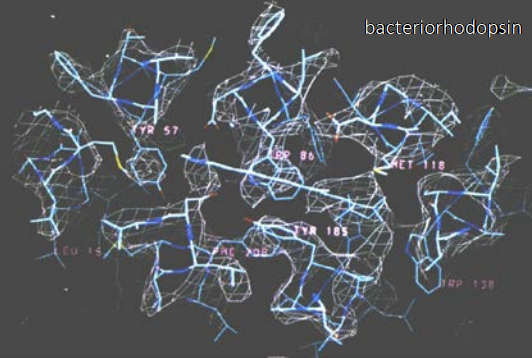
La técnica crio-EM (criomicroscopia electrónica) de alta resolución actual nació en el año 1990, con el trabajo de Henderson y varios colegas (31). Lograron caracterizar a escala atómica una bacteriorodopsina usando cristales bidimensionales enfriados a temperaturas criogénicas. Usando el mismo procedimiento se logró caracterizar otras biomoléculas, como los dímeros de tubulina (1998) y la acuaporina (2000). Estos trabajos pioneros que usaron diferentes microscopios electrónicos de todo el mundo permitieron determinar las características ideales que debería tener dicho instrumento para un crio-EM óptima.
Lo ideal era una fuente de electrones no destructiva de baja intensidad que se pudiera usar en un microscopio electrónico con contraste de fase. Así sería posible determinar la estructura atómica de una biomolécula con un peso molecular de hasta ~50 kDa con una resolución de unos ~3 Å en una muestra formada por unas ~10 000 biomoléculas idénticas situadas en un plano 2D pero cada una con una orientación diferente en 3D.
Había que manipular las moléculas individuales en la muestra para garantizar que sus orientaciones fueran diferentes y se pudieran observar desde todos los ángulos posibles. Frank y varios colegas había desarrollado en los 1970 una técnica para lograrlo con ciertas biomoléculas. A partir de su trabajo pionero Dubochet y otros colegas desarrollaron una técnica para la formación de biopelículas con estas biomoléculas para su uso como muestras en microscopia electrónica. En 1984 se lograron las primeras micrografías electrónicas de virus en suspensión usando estas técnicas de preparación de muestras.

Durante la década de los 1980 el camino fue allanándose para el desarrollo de las técnicas de crio-EM de alta resolución actuales. Los trabajos pioneros de Dubochet, Frank y Henderson han permitido el desarrollo de una nueva herramienta en biología estructural que nos ha ofrecido las maravillosas imágenes que hoy en día decoran todos los libros de texto de Bioquímica y Biología Molecular. Unos 60 años después de los trabajos en cristolografía de John Kendrew y Max Perutz (Premio Nobel de Química de 1962) la técnica de crio-EM ha ofrecido imágenes mucho más allá de lo que podían soñar dichos pioneros.
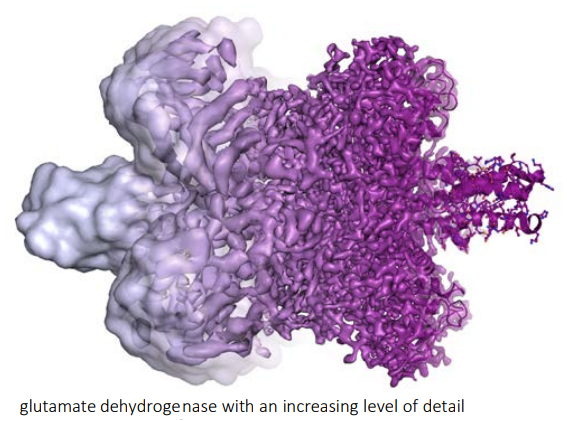
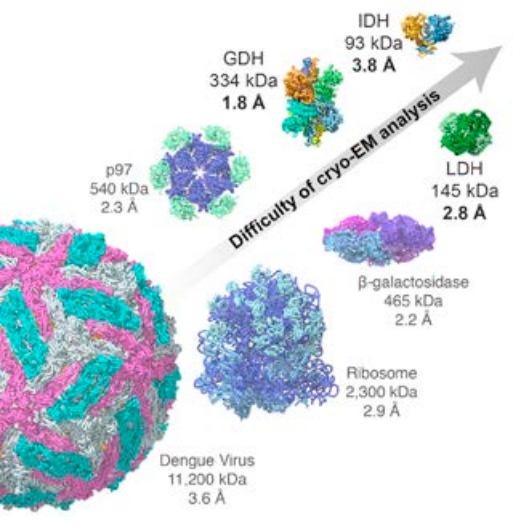
Hoy en día podemos ver con precisión atómica grandes biomoléculas, incluso virus completos y orgánulos celulares. Esta imagen de la proteína glutamato deshidrogenasa (334 kDa) muestra una resolución creciente de izquierda a derecha, hasta alcanzar una resolución máxima de 1,8 Å (combina imágenes obtenidas entre 2012 y 2013). Como en biología molecular la estructura determina la función de las proteínas (enzimas), los avances en crio-EM permiten comprender mucho mejor la fisiología de las células, lo que tiene importantes aplicaciones biomédicas.
Recomiendo leer a Daniel Cressey, Ewen Callaway, «Cryo-electron microscopy wins chemistry Nobel,» Nature News, 04 Oct 2017.



Hola Francis
En la nota decis que con Crio EM podemos ver celulas completas con resolucion atomica? cada biomolecula dentro de la celula completa ? Vi el virus del zika con todas sus proteinas pero nunca una celula completa o una bacteria?
Segunda pregunta, cuales son las tecnicas actuales que se estan investigando, ademas del XFEL y SLAC, que van mas alla de Crio EM?
Gracias, Benjamin, por estar atento; tienes razón, no hay ninguna célula procariota completa que hay sido resuelta por criomicroscopia (aclara la cuestión en la entrada). Por otro lado, hay muchas técnicas aunque las más prometedoras ahora parecen ser XFEL y MicroED. Lo importante es que en las técnicas más recientes no importa la técnica concreta como tal sino la instalación que la soporta; por ejemplo, para XFEL tienes EuXFEL, FERMI Fac., LCLS, PAL, SACLA, SwissFEL, etc. y cada es diferente y complementaria; más información en Vivien Marx, «Structural biology: doors open at the European XFEL,» Nature Methods 14: 843–846 (2017), doi: 10.1038/nmeth.4394.
Hola Francis,
¿Qué aplicación tiene esta técnica?
Me refiero a la capacidad que aporta de visualizar una biomolécula. Y, en general, cuán de importante es la «imagen» per se frente a la posibilidad existente anteriormente de conocer su composición, las reacciones que provoca con otras moléculas, etc.
Y en qué áreas: medicina, industria, etc.
Saludos
Víctor, como la estructura determina la función, conocer la estructura 3D detallada es fundamental en bioquímica; hay varios métodos diferentes y uno de ellos es la criomicroscopia. Sus aplicaciones son «infinitas» (toda aplicación que requiera biomoléculas se beneficia de que conozcamos en detalle su estructura 3D); por tanto, desde la medicina a la industria del petróleo se benefician de este trabajo. Por cierto, te parecerá mentira, pero conocemos dichas estructuras 3D para muy pocas biomoléculas (hay cientos de miles de interés aplicado cuya estructura desconocemos en detalle).
Dices ‘La criomicroscopía electrónica ha permitido ver ciertas biomoléculas con una resolución atómica’ y ‘Hoy en día podemos ver con precisión atómica grandes biomoléculas, incluso virus completos y orgánulos celulares.’ pero la resolución de la CryoEM en 2017 no era atómica (ni ahora salvo contadas ocasiones). Y ¿qué estructura de orgánulos se ha obtenido por CryoEM?
¿En español no sería mas correcto crio-ME que crio-EM?
La estructura de la bacteriorodopsina, los dímeros de tubulina y la acuaporina se determinó mediante la técnica de ‘electron crystallography’ lo que ahora es ‘MicroED’ con cristales en 3D, estas técnicas producen patrones de difracción. El procesamiento de datos con esas técnicas es fundamentalmente distinto del de la CryoEM ‘single particle analysis’.