Las manchas del iris del ojo indican a los iridólogos nuestra predisposición a ciertas enfermedades. Las líneas de la mano indican a los quiromantes algo parecido. La cara es el espejo del alma y muchos afirman ver nuestro estado de salud en nuestra propia faz. Sin embargo, para muchos esto es pseudociencia. ¿Quién dedicaría cientos de millones de euros a estudiar las correlaciones entre estos fenotipos y nuestra salud?
Para la mayoría es obvio, hoy en día, que nuestra predisposición a ciertas enfermedades está escrita en nuestros genes. Para la mayoría es obvio que conocer el ADN de cada uno de nosotros permitirá ahorrar miles de millones de euros a los sistemas sanitarios de los países occidentales. Es por ello que se invierten miles de millones de euro en estudiar técnicas para secuenciar genomas humanos de forma ultrarrápida. Hoy todos tenemos móvil. Muchos creen que en unos años todos llevaremos encima un pequeño chip con nuestro ADN grabado. Y muchos otros creen que estaremos dispuestos a pagar su coste (igual que pagamos lo que cuesta nuestro televisor o nuestro ordenador personal).
El número de genomas humanos secuenciados completamente se acaba de doblar (ya hay 6) como nos cuenta Elizabeth Pennisi, «Number of Sequenced Human Genomes Doubles,» Science, 322: 838, 7 November 2008 . Hace menos de una década costó cientos de millones de dólares secuenciar un único genoma humano. Esta semana se han publicado el primer genoma de un africano, el primero de un asiático y el primero de un enfermo de cáncer. Sólo cuatro genomas publicados de individuos anónimos, aunque ya algunos famosos también tienen su genoma secuenciado como J. Craig Venter y James Watson. Estos genomas han costado entre 250 y 500 mil dólares cada uno. El costo seguirá bajando y varias compañías tienen como objetivo bajarlo a unos «ridículos» 1000 dólares por ADN.
Estos nuevos genomas, todavía a alto coste, son la base para estudios de la variabilidad genética entre poblaciones humanas y para identificar variaciones relacionadas con la predisposición a enfermedades. Especial interés tienen las cambios de un sólo nucleótico (SNP) y las variaciones estructurales en secuencias duplicadas dentro del propio ADN. Actualmente, se conocen millones de estas variantes y su análisis bioinformático está entre las prioridades en el campo de la genética médica.
Richard Wilson y sus colaboradores de la Washington University School of Medicine, en St. Louis, Missouri, han secuenciado los genomas de una célula de un tumor y una célula normal en una mujer que sufre leucemia mielogénica aguda (AML), Timothy J. Ley et al. «DNA sequencing of a cytogenetically normal acute myeloid leukaemia genome,» Nature, 456: 66-72, 6 November 2008 . La diferencia entre ambos genomas es muy pequeña, menor del 3% de los SNP encontrados. Entre todos los SNP encontrados, los investigadores han destacado 10 como «aparentemente» únicos y característicos de estas células tumorales. Wilson afirma, «creo que no se nos ha escapado ninguno más.» Dos aparecen en genes asociados a la leucemia, pero los ocho restantes apuntan a nuevos candidatos a genes asociados con la leucemia. Estudios similares con otros cánceres no tardarán en llegar y prometen revolucionar nuestro conocimiento sobre las bases genéticas de los diferentes (cientos) tipos de cáncer.
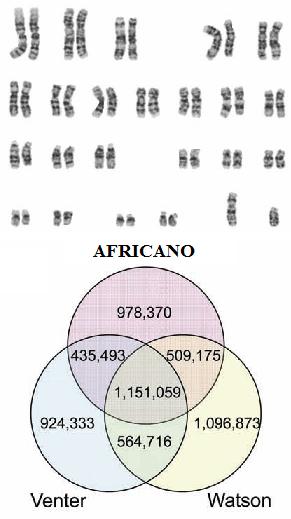
David R. Bentley y sus colaboradores de la empresa británica Illumina Cambridge Ltd. (antes Solexa Ltd.), han secuenciado el genoma de un nigeriano de la tribu de los Yoruba (ver figura), básicamente para demostrar sus avances en la tecnología de secuenciación automática de genomas, David R. Bentley et al. «Accurate whole human genome sequencing using reversible terminator chemistry,» Nature, 456: 53-59, 6 November 2008 . Han necesitado 8 semanas y unos 250.000 dólares. Lo que no es mucho. Las tecnologías de secuenciación masivamente paralela permitirán convertir la secuenciación del genoma en una herramienta clínica imprescindible en el futuro, proclaman los autores, obviamente mostrando que son parte interesada en ello.
Jiang Wang del Beijing Genomics Institute, en Shenzhen, China, y sus colegas han secuenciado el genoma de un hombre chino de Han, Jun Wang et al. «The diploid genome sequence of an Asian individual,» Nature, 456: 60-65, 6 November 2008 . Mientras que el africano mostraba unos 4 millones de SNP, de los cuales 1 millón eran nuevos, el genoma del chino muestra unos 3 millones de los cuales 417.000 son nuevos. Como es de esperar, la variación genética por kilobase del africano es mayor que la del chino y que la del caucásico (secuenciado por el Proyecto Genoma Humano, Nature, 2001, y por Celera Genomics, Science, 2001), dado que sus ancestros se remontan más lejos en la escala evolutiva humana.
La empresa Illumina espera que el costo de secuenciar un genoma baje a unos 10.000 dólares el año que viene y a unos 1000 dólares en un lustro. La competencia entre varias empresas del sector a nivel mundial garantizará que cumplan con sus objetivos. El problema ahora es otro, ¿cómo interpretar clínicamente los datos ofrecidos por un genoma dado? ¿Para qué sirve conocer un genoma? Ahora mismo, para lo mismo que sirve la quiromancia (lectura de las manos) o la iridología (lectura del iris) o la intución de nuestra abuela («niño qué mala cara tienes»). Ahora mismo, para nada. Dentro de unas décadas, la mayoría espera que sirva para mucho. ¿Se cumplirán sus expectativas?

