
Ya puedes disfrutar del episodio 6 del podcast Biosíntesis [Medium, iVoox, iTunes, Google podcasts, Spotify, TuneIn, Radio Public, PlayerFM]. El episodio está fechado el 24 de mayo de 2019, aunque se publica hoy; fue grabado en el estudio de radio de la Facultad de Ciencias de la Comunicación de la Universidad de Málaga (UMA). Presentamos artículos científicos sobre bioquímica y biología molecular bajo la batuta de José (Pepe) Lozano @leviaingenia (Dep. Biología Molecular y Bioquímica, UMA), participamos Silvana Tapia @SilvanaTapia3 (Dep. Microbiología , UMA), y un servidor, Francisco R. (Francis) Villatoro, @emulenews (Dep. Lenguajes y Ciencias de la Computación, UMA). Así como bionoticias y anuncios gracias a Belén Delgado Martín, @bio_Belen (Graduada en Bioquímica, UMA) e Íker Puerto San Román, @SrMitocondrio (estudiante del Grado de Bioquímica, UMA), autor del blog La Mitocondria.
En nuestra sección de reseñas hemos reseñado el libro «Virus» de José Antonio López (JAL) [LCMF, 02 jun 2019], incluyendo una apasionante entrevista al autor.

Tras un breve homenaje de Pepe a Bob Dylan, Premio Nobel de Literatura de 2016, Silvana nos presenta un artículo sobre biología sintética. Se ha sintetizado el genoma (ADN de 4 megapares de bases) de la bacteria Escherichia coli con dos cambios en la codificación (es decir, se ha modificado el código genético en el genoma sintético). La síntesis de proteínas usa 64 codones para codificar los 20 aminoácidos y la parada (Stp) de la transcripción. La serina (Ser) está codificada por 6 codones en el genoma natural y se ha recodificado con 4 codones en el artificial (dejando libres los codones TCG y TCA); el codón de parada (Stp) está codificado por 3 codones y se ha recodificado con 2 codones (dejando libre el codón TAG). Así el genoma artificial solo usa 61 codones para codificar los 20 aminóacidos y la parada (este proceso ha requerido recodificar 18 214 codones).

Además se ha realizado una refactorización para eliminar algunos solapes entre marcos de lectura abiertos (ORF), lo que ha conducido a que el genoma sintético sea algo más largo que el natural. La recodificación y refactorización del genoma no impide que la bacteria sea totalmente funcional (en apariencia), aunque parece que su crecimiento se ha penalizado un poco y resulta más lento (pero no se sabe el porqué). La ventaja de disponer de 3 codones no usados es que permitirá usarlos para codificar nuevos aminóacidos que puedan tener interés industrial y/o farmacéutico. Silvana ha entrevistado a uno de los autores, Daniel de la Torre, que nos comenta en primera persona detalles muy interesantes de su trabajo. El artículo es Julius Fredens, …, Daniel de la Torre, …, Jason W. Chin, «Total synthesis of Escherichia coli with a recoded genome,» Nature 569: 514-518 (15 May 2019 ), doi: 10.1038/s41586-019-1192-5; recomiendo también Benjamin A. Blount, Tom Ellis, «Construction of an Escherichia coli genome with fewer codons sets records,» Nature 569: 492-494 (16 May 2019), doi: 10.1038/d41586-019-01584-x.

Yo he comentado dos artículos publicados en Nature y Science que presentan nuevas estrategias antitumorales basadas en la reactivación de las proteínas RB y PTEN, dos supresores tumorales bien conocidos. Los genes supresores de tumores, y las proteínas que expresan, están implicados en los mecanismos de control que regulan la división celular. Los tumores desactivan estos genes y proteínas. Se están desarrollando nuevas terapias contra el cáncer basadas en reactivar estas proteínas supresoras de tumores. En Nature se proponen nuevas dianas terapéuticas para reactivar la proteína supresora de tumores Rb (retinoblastoma) en cáncer de pulmón; en Science se centran en la proteína supresora de tumores PTEN en cáncer de próstata. Sin lugar a dudas, las nuevas terapias que auguran estos estudios son muy prometedoras y se podrán extender a otros tipos de cáncer.
En el desarrollo del cáncer cambia la actividad de muchos oncogenes y supresores de tumores mediante la acción de interruptores enzimáticos que los fosforilan (quinasas) o desfoforilan (fosfatasas). Se ha prestado más atención a la inhibición terapéutica de las quinasas oncogénicas que se activan por mutación. El supresor de tumores PTEN (homólogo de fosfatasa y tensina en el cromosoma 10) es la fosfatasa inactivada más frecuente en el cáncer humano. PTEN suprime el crecimiento tumoral ejerciendo un efecto antagonista en las proteínas de la ruta de señalización PI3K/AKT (fosfatidil inositol 3 quinasa), una de las más importantes implicadas en el cáncer.
El primer artículo es David M. Walter, Travis J. Yates, Miguel Ruiz-Torres, …, David M. Feldser, «RB constrains lineage fidelity and multiple stages of tumour progression and metastasis,» Nature 569: 423-427 (16 May 2019), doi: 10.1038/s41586-019-1172-9; más información en Seth M. Rubin, Julien Sage, «Manipulating the tumour-suppressor protein Rb in lung cancer reveals possible drug targets,» Nature 569: 343-344 (16 May 2019), doi: 10.1038/d41586-019-01319-y. El segundo artículo es Yu-Ru Lee, Ming Chen, …, Pier Paolo Pandolfi, «Reactivation of PTEN tumor suppressor for cancer treatment through inhibition of a MYC-WWP1 inhibitory pathway,» Science 364: eaau0159 (17 May 2019), doi: 10.1126/science.aau0159; más información en Ramon Parsons, «Restoring tumor suppression,» Science 364: 633-634 (17 May 2019), doi: 10.1126/science.aax5526.
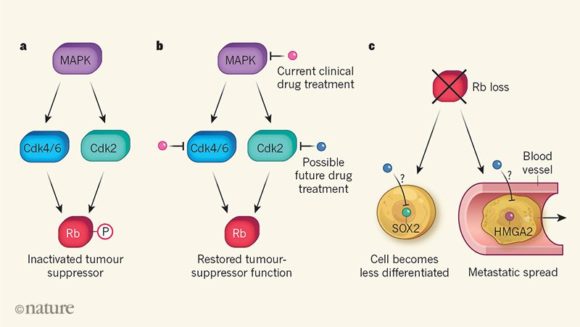
El retinoblastoma es un cáncer ocular en niños pequeños que se desarrolla en la retina por una mutación en la proteína Rb, codificada por un gen supresor de tumores llamado RB1. La proteína Rb actúa como un freno para la progresión del ciclo celular y promueve la diferenciación celular en un tipo específico; muchos tumores humanos bloquean o desactivan esta proteína mediante unas enzimas llamadas quinasas dependientes de ciclina (CDK), que fosforilan a Rb (añaden grupos fosfato). Ya hay tratamientos antitumorales que usan fármacos inhibidores de la actividad de las quinasas Cdk4 y Cdk6, impidiendo que inactiven a Rb. También los hay inhibiendo la cascada MAPK (formada por tres proteínas quinasas activadas por mitógenos) que regula a nivel postranscripcional a las CDK.
Sin embargo, estos tratamientos no son eficaces en muchos tumores, por lo que se requiere el descubrimiento de nuevas dianas terapéuticas. Ese es el objetivo del grupo de David M. Feldser, del Department of Cancer Biology, Perelman School of Medicine, University of Pennsylvania, en Philadelphia, EE.UU., autor principal del artículo publicado en Nature, cuyos tres primeros autores (que han contribuido por igual) son David M. Walter, Travis J. Yates y Miguel Ruiz-Torres miembros del grupo de Feldser. Usando un modelo en ratón del cáncer de pulmón han descubierto el rol de la quinasa Cdk2 en la inactivación de Rb. Así proponen futuros fármacos que actúen inhibiendo la quinasa Cdk2. Se espera que el desarrollo de medicamentos dirigidos a esta proteína sea prometedor en el tratamiento del cáncer de pulmón.
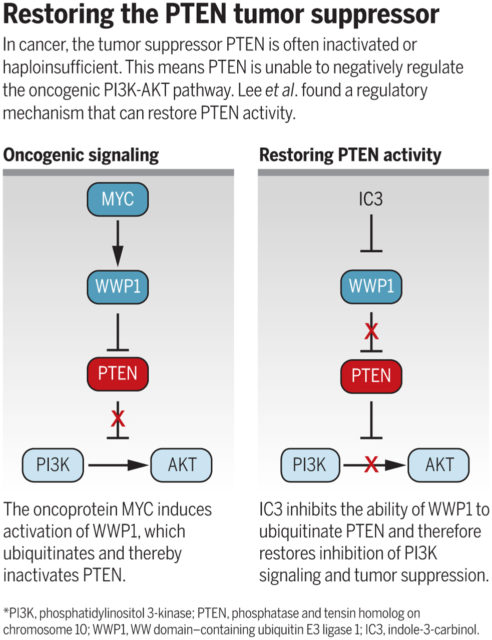
PTEN es un gen supresor de tumores antagonista de la ruta de señalización oncogénica (PI3K)–AKT. Las células cancerosas no pueden perder la actividad completa de PTEN de forma prematura, porque se dispararía la senescencia celular. Por esta razón, la desactivación de PTEN ocurre un proceso de varias etapas. El nuevo trabajo en Science estudia diferentes vías de activación y desactivación de PTEN. Un cribado para detectar proteínas que interaccionan con PTEN identificó la tercera actividad (E3) de una ubiquitina ligasa WWP1 con un dominio de tipo HECT; en concreto, realiza una poliubiquitinación de PTEN lo que inhibe su actividad como fosfatasa y activa la ruta de señalización AKT. La ubiquitina es un polipéptido de 76 aminoácidos que puede formar polímeros, cadenas de poliubiquitinas, que son señales de proteólisis (destrucción de proteínas). La poliubiquitinación de PTEN disparada por WWP1 suprime su dimerización en la membrana que permite su función supresora de tumores. Muchos cánceres tienen una expresión amplificada de WWP1.
Resulta que la expresión (transcripción) de WWP1 es activada por la oncoproteína MYC, que suprime la actividad de PTEN y fomenta la ruta de señalización PI3K-AKT. Un cribado de productos naturales en vegatales descubrió que I3C (indole-3-carbinol) es un inhibidor de WWP1 y la ubiquitinación de PTEN mediada por el dominio HECT de WWP1; así disminuye la señalización AKT en células de cáncer de próstata, reduciendo el crecimiento tumoral (aunque cánceres que carecen de PTEN, en los demás se espera que WWP1 sea beneficiosa). Por ello se propone su uso como fármaco para reactivar PTEN. Este resultado se ha publicado en Science por el grupo de Pier Paolo Pandolfi, del Cancer Research Institute, Beth Israel Deaconess Cancer Center, Harvard Medical School, Boston, MA 02215, USA, y del Department of Medicine and Pathology, Beth Israel Deaconess Medical Center, Harvard Medical School, Boston, MA 02215, USA; los dos primeros autores son miembros de su grupo, Yu-Ru Lee y Ming Chen. Por supuesto, se requiere una mayor investigación de la regulación de PTEN mediada por WWP1.

Tras las bionoticias, Pepe ha entrevistado al investigador Manuel Collado (CHUS, Santiago de Compostela) quien nos comenta su artículo en el que estudia la doble función oncogénica y de reprogramación a pluripotencia del oncogén RAS, ambas funciones son dependientes del contexto celular. La reprogramación celular y la transformación oncogénica comparten características comunes; ambas implican un gran cambio en la identidad celular. El nuevo trabajo indica que RAS induce cambios en la expresión que promueven la pérdida de la identidad celular y la adquisición de la entidad madre de manera paracrina. Estos cambios resultan en la reprogramación cuando se combinan con factores de reprogramación.
El resultado podría tener aplicaciones terapéuticas dado que, entre células portadoras de defectos oncogénicos que cooperan, RAS lleva dichas células a un destino celular incompatible con la malignidad. El artículo es Alba Ferreirós, Pablo Pedrosa, …, Manuel Collado, «Context-Dependent Impact of RAS Oncogene Expression on Cellular Reprogramming to Pluripotency,» Stem Cell Reports 12: 1099-1112 (14 May 2019), doi: 10.1016/j.stemcr.2019.04.006.

En la sección de (bio)noticias, Íker nos comenta un nuevo avance sobre las bases de los trastornos del espectro autista publicado en Science (Dmitry Velmeshev, Lucas Schirmer, …, Arnold R. Kriegstein, «Single-cell genomics identifies cell type–specific molecular changes in autism,» Science 364: 685-689 (17 May 2019), doi: 10.1126/science.aav8130). «El autismo en sí no es un único trastorno, sino toda una serie de trastornos que se encuadran dentro del llamado espectro autista (TEA). Estos trastornos son muy heterogéneos desde el punto de vista clínico y a nivel genético, pues en estos están implicados entre 600 y 1200 genes diferentes, cuyas interacciones entre sí se desconocen, al igual que su implicación en la mayor o menor gravedad de los síntomas presentados por los pacientes. Estudios anteriores sobre la expresión génica en pacientes con autismo habían encontrado que determinados cambios en la neocorteza cerebral se debían a alteraciones de ciertos genes y rutas metabólicas».
«El equipo del Dr. Kriegsten (Universida de California, San Francisco) ha estudiado el llamado transcriptoma de ciertos tipos celulares en 41 muestras postmortem de corteza prefrontal y de córtex del cíngulo anterior de 15 pacientes con TEA y 16 individuos de control, todos entre los 4 y 22 años de edad. A ninguno de estos pacientes se les había diagnosticado discapacidad intelectual, pero la mitad de los pacientes de autismo si habían padecido convulsiones, un síntoma común de estos trastornos. Por tanto, con el objetivo de hacer una comparación, también se usaron muestras de pacientes con epilepsia esporádica. Mediante el aislamiento de los núcleos celulares de las muestras, obtuvieron 104.600 perfiles de expresión e identificaron 17 tipos celulares diferentes. Cuando se compararon los perfiles de expresión de los pacientes de TEA con los de los controles se hallaron 692 eventos de expresión diferentes y 513 genes diferencialmente expresados. Los principales fenómenos celulares afectados por estas alteraciones son la transmisión sináptica, la orientación de los axones, la migración neuronal y la señalización del ácido γ-aminobutírico (GABA). También se hallaron alteraciones en la expresión de factores de transcripción importantes en el desarrollo del cerebro, como SOX5 y TCF25».
«Los principales tipos celulares afectados por estas alteraciones fueron las neuronas excitatorias L2, L3 y L4 y las células de microglía, entre otros. Este equipo también descubrió que existe una correlación entre la gravedad de las alteraciones presentes en las neuronas L2 y L3 y en las células de microglía con la severidad del trastorno del paciente. En cuanto a las muestras de pacientes con epilepsia espontánea, se halló que compartían un 10% de las alteraciones en la expresión génica observadas en los pacientes con autismo, lo que nos permite diferenciar qué genes están detrás de las convulsiones y cuáles están detrás de la patogénesis de los TEA. Estudios como este son muy importantes, pues el estudio de las bases genéticas y moleculares detrás de enfermedades, en este caso los TEA, nos permiten identificar posibles dianas terapéuticas. Estudios con un mayor número de muestras y un análisis más exhaustivo permitirán hallar con más precisión las causas genéticas de los TEA y plantearán posibles tratamientos. Estaremos atentos».

La bionoticia que Belén nos trae está basada en un News & Views en Nature (Simon T. Schafer, Fred H. Gage, «Nerve cells from the brain invade prostate tumours,» Nature 569, 637-638 (15 May 2019), doi: 10.1038/d41586-019-01461-7, sobre el artículo de Philippe Mauffrey, Nicolas Tchitchek, …, Claire Magnon, «Progenitors from the central nervous system drive neurogenesis in cancer,» Nature 569: 672-678 (15 May 2019), doi: 10.1038/s41586-019-1219-y). «Muchos estudios en la última década han mostrado que la interacción entre las células tumorales y las células nerviosas puede influir en la progresión de varios tipos de cáncer. En los tumores de próstata, la presencia de nuevas células nerviosas está asociada a una mayor malignidad y capacidad metastásica. El microambiente tumoral permite el desarrollo de estas células en un proceso llamado neo-neurogénesis, pero no se conoce el origen inicial de estas células nerviosas en los tumores. Mauffrey y colaboradores, del Instituto de Biología François Jacob, de París, publican en Nature que las células progenitoras neurales de una zona en la que se da neurogénesis en el cerebro son capaces de migrar hacia los tumores de próstata, invadiendo el microambiente tumoral y dando lugar a la formación de nuevas células nerviosas».
«El modelo usado se llama Hi-myc. Los autores observaron que los tumores de próstata en estos ratones estaban asociados a unas células nerviosas llamadas neuroblastos, que expresan una proteína llamada doblecortina (DCX), también expresada en células progenitoras de neuronas. Aislaron las células que expresaban DCX y analizaron la expresión de genes y proteínas. No expresaban Myc, un oncogén driver en tumores de próstata, por lo que se descartó que su origen fuera a partir de las células tumorales. Se observaron características moleculares parecidas a las de las células progenitoras neurales y podían diferenciarse in vitro en células parecidas a las neuronas. Después, rastrearon la procedencia de las células usando una técnica de marcaje en el cerebro, revelando que las células progenitoras de la zona subventricular son capaces de migrar hacia la localización de los tumores de próstata. Determinaron la relevancia de sus hallazgos en otros tipos de cáncer usando otros modelos trasplantados con células tumorales humanas, observándose una forma similar de migración de las células progenitoras neurales hacia los tumores».
«Sin embargo, en el cerebro humano, existe un consenso respecto a que se da neurogénesis en el giro dentado, pero es necesario determinar si esto ocurre también en la zona subventricular, de donde proceden las células nerviosas que se establecen en el tumor. Esto implica que serán necesarios nuevos estudios para determinar si este mecanismo tiene relevancia clínica en humanos. Además, no se puede descartar por completo la posibilidad de que se formen células nerviosas a partir de otras células tumorales, ya que el modelo utilizado quizá no sirve para todos los tipos de cáncer de próstata».
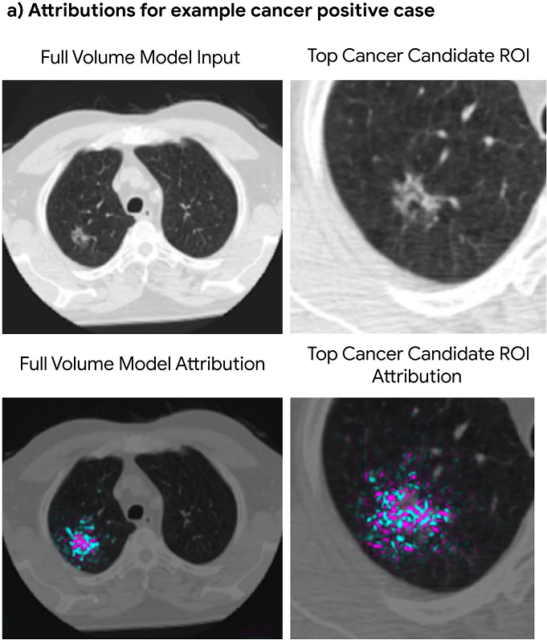
Íker se hace eco de la noticia «Baja la mortalidad por melanoma por primera vez en España», El Mundo, 07 may 2019. «La mortalidad por melanoma en menores de 65 años está disminuyendo por primera vez en España». Mientras que Belén lo hace de la noticia de Daniel Mediavilla, «Las máquinas empiezan a ganar a los humanos en la predicción del riesgo de cáncer», Materia, El País, 20 may 2019, sobre el artículo de Diego Ardila, Atilla P. Kiraly, …, Shravya Shetty, «End-to-end lung cancer screening with three-dimensional deep learning on low-dose chest computed tomography,» Nature Medicine 25: 954-961 (20 May 2019), doi: 10.1038/s41591-019-0447-x. «Un algoritmo diseñado por Google y la Universidad del Noroeste en EE UU obtiene mejores resultados que los radiólogos analizando tomografías en busca de indicios de tumores».
Además, Belén y Silvana nos presentan Sciencebook, una nueva plataforma social para divulgar la ciencia, y entrevistan a su fundador Sergio Marcuello. Más información en Lydia Gil, «ScienceBook, la red social de investigación y divulgación marca España», Social Media en Investigación, 13 mar 2019.

«La sección El Libro de la Semana nos trae la última obra del conocido divulgador José Antonio López, más conocido como JAL (responsable del programa radiofónico «Entre probetas» de RNE). Se trata del libro «Virus. Ni vivos ni muertos» (ed. Almuzara), en el que el autor da un repaso bastante completo al catálogo de los principales virus que nos afectan a los humanos, sus aspectos moleculares y la historia de los variados y numerosos esfuerzos que los científicos han realizado a lo largo de la historia por luchar contra las enfermedades que han causado. Un libro, sin duda, muy recomendable, por su rigor y por lo ameno de su lectura. En el episodio charlamos largo y tendido con el autor, JAL, sobre virus en general y los descritos en el libro, más en particular». En este blog puedes leer mi «Reseña: «Virus» de José Antonio López (JAL)», LCMF, 02 jun 2019.
¡Qué disfrutes del podcast!

