
HAL, la inteligencia artificial de la película ‘2001: una odisea del espacio’, ha inspirado el nombre de un nuevo antibiótico, la halicina. Se ha descubierto gracias a un algoritmo de aprendizaje automático (deep learning). Se publican en Cell pruebas que apuntan a que es uno de los antibióticos más potentes conocidos. En ratones se ha demostrado su actividad actibacteriana contra superbacterias resistentes a antibióticos, como Clostridium difficile y Acinetobacter baumannii, así como otras bacterias de gran interés biomédico, como Mycobacterium tuberculosis y varias cepas de Enterobacteriaceae. La inteligencia artificial ha descubierto otros candidatos a antibióticos, pero aún están en estudio.
El trabajo está liderado por Regina Barzilay y James J. Collins, ambos en el MIT (Boston, EEUU). Se partió de una base de datos de 2560 moléculas (2335 sin repeticiones), de las que 232 (7.97%) mostraban actividad antibiótica; se entrenó una red neuronal “profunda” (deep learning) con dicha base de datos para que aprendiera a correlacionar la estructura atómica y molecular (representada en forma de grafo tridimensional) con su actividad antibacteriana contra Escherichia coli (determinada en laboratorio). Luego se usó esta inteligencia artificial para explorar una base de datos de 6111 moléculas candidatas a fármacos para humanos (en diferentes estados de investigación). Así se descubrió la actividad antibiótica de SU3327, rebautizado halicina, un fármaco que se desarrolló para tratar la diabetes, pero que se descartó antes de llegar a la práctica clínica. Usando experimentos de laboratorio con ratones se descubrió que era un potente antibiótico de amplio espectro (a pesar de que su estructura química es muy diferente a la de otros antibióticos).
El algoritmo se ha usado para explorar 107 millones de compuestos de la base de datos ZINC15 (que contiene ∼1.5 miles de millones); se han descubierto 23 antibióticos potenciales; entre ellos destacan dos que parecen muy prometedores (ZINC000100032716 y ZINC000225434673). Su estudio experimental en laboratorio está en curso. Sin lugar a dudas, la búsqueda automática de fármacos y antibióticos mediante herramientas de inteligencia artificial revolucionará la biomedicina. Por ello habrá que estar al tanto de sus futuros éxitos. El artículo pionero es Jonathan M. Stokes, Kevin Yang, …, James J. Collins, ”A Deep Learning Approach to Antibiotic Discovery,” Cell 180(4): P688–702.E13 (20 Feb 2020). doi: https://doi.org/10.1016/j.cell.2020.01.021
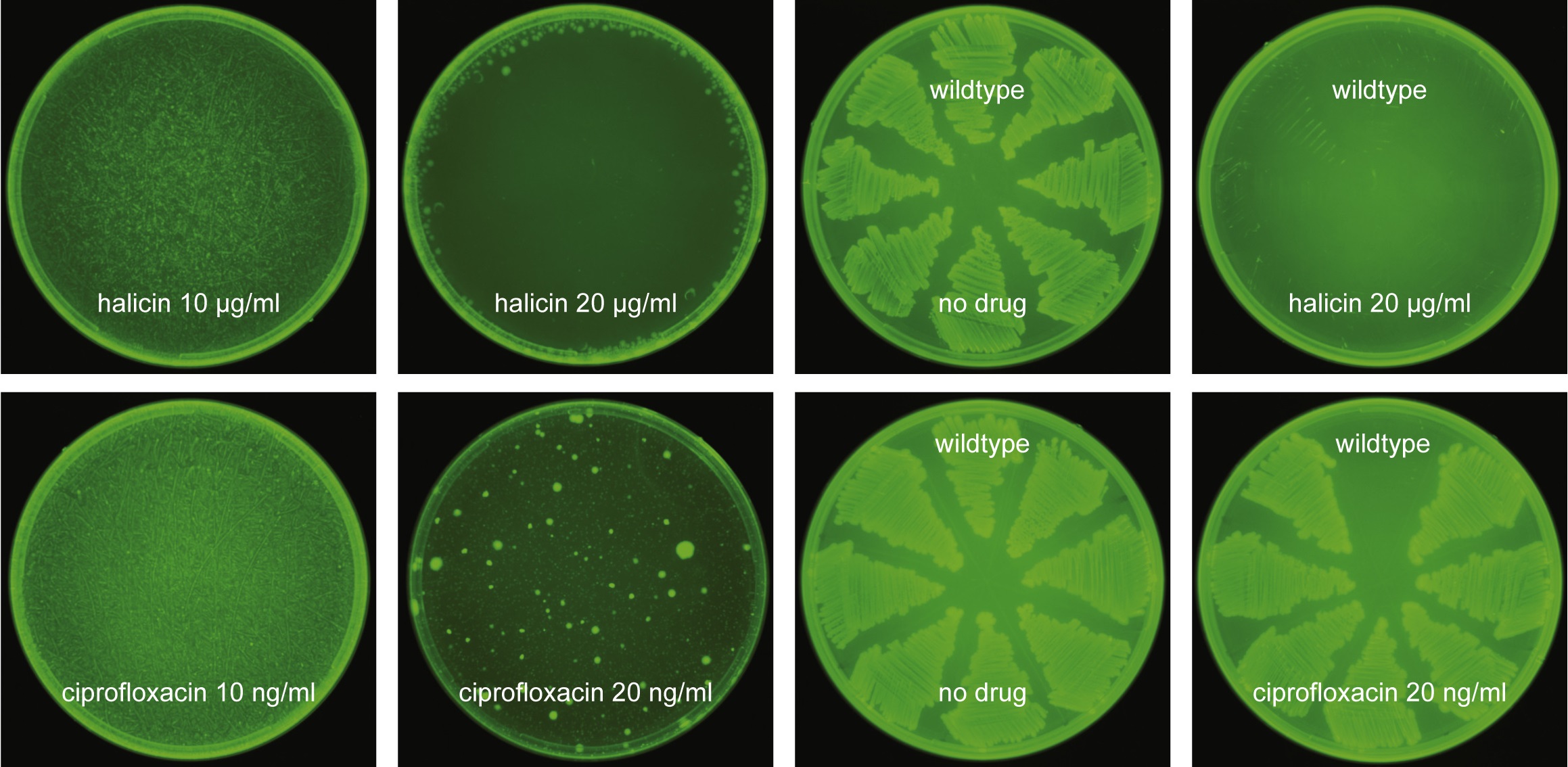
El descubrimiento de nuevos antibióticos es imprescindible para combatir la resistencia bacteriana. Pero los métodos convencionales son muy costosos, lo que desincentiva al sector privado. Además, la mayoría de los nuevos antibióticos son muy similares entre sí a nivel químico. La búsqueda automática de fármacos en bases de datos de moléculas parece un camino prometedor, casi imprescindible. Para ello se necesitan enfoques novedosos, como el aprendizaje automático (machine learning). Por supuesto, la idea no es nueva, y se llevan décadas investigando en quimioinformática para la predicción de propiedades y funciones moleculares.
El aprendizaje profundo está de moda, por eso se ha empezado a usar en la búsqueda de antibióticos desde hace un par de años. Para que la metodología sea exitosa, los modelos de redes neuronales in silico deben combinarse con enfoques analíticos y experimentales in vivo. En el nuevo artículo se ha aplicado un modelo de red neuronal profunda para predecir la inhibición del crecimiento de Escherichia coli usando un conjunto de 2335 moléculas del Drug Repurposing Hub (una base de datos cuyo objetivo es el reposicionamiento de fármacos). La halicina (antes llamada SU3327) es un inhibidor de la proteína quinasa N-terminal de c-Jun (JNK); esta sustancia ha resultado ser un potente inhibidor del crecimiento de E. coli aunque tiene una estructura molecular muy diferente a otros antibióticos conocidos. La investigación in vitro y con modelos murinos in vivo ha revelado que la halicina tambén es un potente inhibidor del crecimiento de un amplio espectro de patógenos.

Los detalles del algoritmo de aprendizaje de profundo usado se pueden consultar en el artículo publicado en Cell (que es de acceso gratuito). El algoritmo seleccionó 99 moléculas entre las 2335 de la base de datos de búsqueda. Se probaron en laboratorio y se confirmó que 51 de ellas inhibían el crecimiento de E. coli. Las puntuaciones más altas según la inteligencia artificial correlacionan con una mayor probabilidad de inhibición del crecimiento; entre las 63 moléculas con puntuaciones más bajas solo 2 mostraban actividad inhibidora del crecimiento. Se entrenó una segunda red neuronal profunda para que identificara la toxicidad de estas moléculas. Se aplicó a las 51 moléculas para buscar la que presentaba menor toxicidad. Así relució la halicina cual pepita de oro. Pero solo se había logrado el primer paso; el siguiente era demostrarlo en laboratorio.
Los estudios clínicos mostraron que la halicina es un antibiótico bactericida de amplio espectro. Como su estructura molecular es muy diferente a la de otros antibióticos, se cree que podría funcionar a través de un mecanismo de acción poco común. La hipótesis de los autores es que disipa el gradiente de protones (ΔpH) a través de la membrana, lo que penaliza la función de algunas proteínas transmembrana e inhibe el crecimiento. Los estudios in vitro parecen confirmar esta hipótesis, aunque hay que seguir investigando en los detalles del mecanismo. Así, lo autores proponen que la halicina podría esquivar los mecanismos de resistencia bacteriana más comunes.

La inteligencia artificial ha sido aplicada a un conjunto más grande de moléculas, 107 349 233 moléculas de la base de datos ZINC15. Tras 4 días de cómputo se obtuvieron 6820 moléculas con puntuación mayor de 0.7, y 1070 moléculas con mayor de 0.9. Se compararon (usando la métrica de semejanza molecular de Tanimoto) con todos los antibióticos conocidos, con el objetivo de identificar las moléculas antibacterianas con una estructura lo más diferente posible a ellos. Así se identificaron 23 compuestos que eran inhibidores de E. coli; se estudió si también inhibían el crecimiento de alguna otra bacteria (al menos una entre S. aureus, Klebsiella pneumoniae, A. baumannii, y P. aeruginosa); así se redujo el conjunto a solo 8 moléculas. Dos de estas moléculas, ZINC000100032716 y ZINC000225434673, mostraron una potente actividad inhibidora de amplio espectro. Su estudio detallado in vivo en modelos murinos está en curso.
En resumen, el nuevo artículo es solo una prueba de concepto; no sé si la halicina acabará llegando a la clínica, espero que sí. Para mí lo más relevante de este nuevo trabajo es que sugiere que la predicción automática de nuevos fármacos mediante inteligencias artificiales podría revolucionar el campo de la farmacología. Por supuesto, el aprendizaje automático no es la panacea para todo; su utilidad depende del conjunto de entrenamiento seleccionado, así como de otros detalles computacionales. Aún así, los avances en la quimioinformática aplicada a la biomedicina me parecen apasionantes.


Excelente artículo, estoy deseando que funcionen las pruebas clínicas.
¿Alguien más se ha dado cuenta lo revolucionario que es la «fabricación» de antibióticos «in silico»?
Por otro lado las multinacionales se pelearán por tiempo de computación de los superordenadores más que por unos laboratorios competentes. O quizás les abra los ojos para explorar en nuevas moléculas.
Un salto de gigante.
Y todavía falta que entre en acción la computación cuántica con algoritmos más avanzados y eficientes
HALucinante.
La «métrica de semejanza molecular de Tanimoto» es solo la aplicación de la distancia de Tanimoto a los vectores con los que representan las moleculas (vectores de radio 2 y huella de 2048 bits segun el paper).
La distancia de tanimoto es la extension de la distancia de Jaccard para vectores a valores reales.
T(v1,v2) = v1.v2 / ( |v1| ^ 2 + |v2|^2 – v1.v2 )
Donde v1 y v2 son vectores, v1.v2 es el producto interno de ambos y |v1| es la norma del vector.
https://en.wikipedia.org/wiki/Jaccard_index
https://en.wikipedia.org/wiki/Jaccard_index#Tanimoto_similarity_and_distance
Maravilloso como siempre Francis!
Yakko
Gracias, Yakko.
Buenas Tardes. Me interesa el artículo o si están haciendo pruebas experimentales. Tengo mi padre que sospecho se beneficiaria de eso. Soy de ecuador y me gustaría me contacten
tiene que ver con el compuesto natural alicina ( del ajo), con propiedades también antibióticas?
No tiene nada que ver.
la actividad de 1mgr. de alicina equivale a 15 unidades de penicilina, siendo activa sobre los germenes a diluciones tan altas como una parte entre 250.000. La alicina del ajo actúa sobre un amplio abanico de microorganismos como E.coli,
Proteus sp., estreptococos, bacilo piociánico, colibacilos, vibrión colérico, mycobacterium tuberculosis, también es activa sobre estafilococos, rickettsias, shigellas y brucelas…
Alvar, la alicina (S-Alil-2-propentiosulfinato, C6H10OS2) no tiene nada que ver con la halicina (C5H3N5O2S3).
Fascinante. Como químico, estos artículos me fascinan.
gracias, un saludo.
Buenas. Es ya un hecho y se puede tomar la halicina? Tengo a mi mama con esas bacterias y la está pasando muy mal
No, Tete, lo siento. Aún no se han realizado los ensayos clínicos necesarios para verificar su seguridad y su eficacia. Supongo que tardarán varios años en realizarse. Si fueran un éxito, su uso sería aprobado y sería posible su uso.