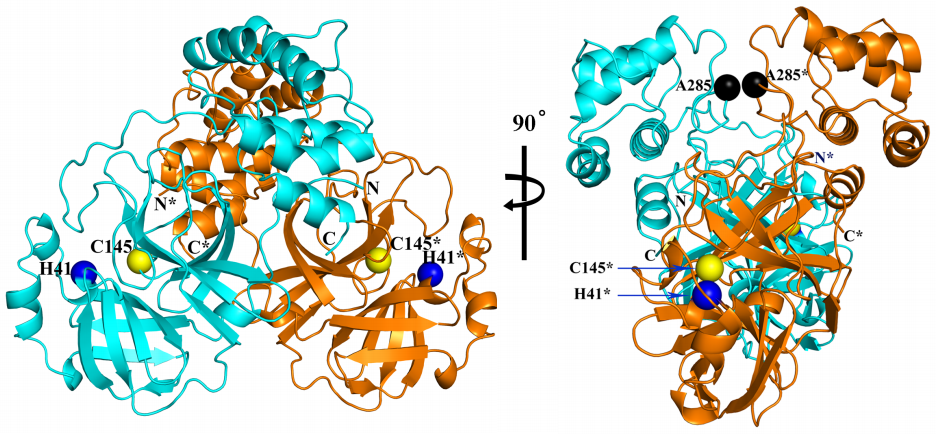
El ARN de los coronavirus codifica poliproteínas que se escinden en varias proteínas responsables de su replicación en la célula huésped. Las proteasas que producen la escisión son dianas terapéuticas preferentes para los fármacos antivirales. La proteasa principal (Mpro), también llamada proteinasa tipo-3C (3CLpro), del coronavirus SARS-CoV-2 se encuentra en la poliproteína ORF1ab. Se ha publicado la estructura tridimensional de este dímero con una resolución de 1.75 Å usando cristalografía de rayos X. Se confirma su gran semejanza con la de SARS-CoV (que se publicó en 2003), como predecían los modelos teóricos basados en homología (la gran semejanza entre sus secuencias de aminoácidos). Además, se ha usado esta estructura para buscar nuevos antivirales usando herramientas bioinformáticas.
Solo 12 aminoácidos diferencian la proteasa principal de SARS-CoV-2 (COVID-19) y la de SARS-CoV (SARS); parece que dos de ellas, A285T y L286I, están relacionadas con una capacidad de infectar 3.6 mayor para el nuevo coronavirus. El cambio A285T permite que dos dominios se aproximen un poco más entre sendas proteínas del dímero; en concreto, la distancia entre los átomos de carbono de los residuos en la posición 285 se encuentran a 6.77 Å en la proteína Mpro de SARS-CoV mientras se acercan hasta 5.21 Å en Mpro de SARS-CoV-2; así los centros de los dominios a los que pertenecen se acercan de 33.4 Å hasta 32.1 Å. Parece que esta diferencia tiene gran relevancia en la búsqueda de antivirales más eficaces.
Se está publicando tanto sobre el nuevo coronavirus (más de 900 artículos, de los que unos 300 están aceptados en revistas con revisión por pares) que estar al tanto de los progresos raya lo imposible. Pero debemos ser optimistas, toda esta investigación acabará dando sus frutos y el próximo año tendremos antivirales y vacunas disponibles contra COVID-19. El artículo con la estructura 3D es Linlin Zhang, Daizong Lin, …, Rolf Hilgenfeld, «X-ray Structure of Main Protease of the Novel Coronavirus SARS-CoV-2 Enables Design of α-Ketoamide Inhibitors,» bioRxiv preprint (20 Feb 2020), doi: https://doi.org/10.1101/2020.02.17.952879; el artículo previo con la de SARS-CoV es Haitao Yang, Maojun Yang, …, Zihe Rao, «The crystal structures of severe acute respiratory syndrome virus main protease and its complex with an inhibitor,» PNAS 100: 13190-13195 (11 Nov 2003), doi: https://doi.org/10.1073/pnas.1835675100. El artículo sobre el posible efecto del A285T en la infectividad es Jianxing Song, «2019-nCoV 3C-Like Protease carries an activity-enhancing T285 /A variation which may contribute to its high infectivity,» OSF Preprints (17 Feb 2020), doi: https://doi.org/10.31219/osf.io/skznv.
En esta pieza también cito dos predicciones teóricas previas: Martin Stoermer, «Homology Models of Coronavirus 2019-nCoV 3CLpro Protease,» chemRxiv preprint (05 Feb 2020), doi: https://doi.org/10.26434/chemrxiv.11637294; y Yu Wai Chen, Chin-Pang Yiu, Kwok-Yin Wong, «Prediction of the 2019-nCoV 3C-like Protease (3CLpro) Structure: Virtual Screening Reveals Velpatasvir, Ledipasvir, and Other Drug Repurposing Candidates,» chemRxiv preprint (10 Feb 2020), doi: https://doi.org/10.26434/chemrxiv.11831103 (F1000 Research). La mayor infectividad asociada al cambio A285T (Ala285Thr) se publicó en Jianxing Song, «2019-nCoV 3C-Like Protease carries an activity-enhancing T285 /A variation which may contribute to its high infectivity,» OSF Preprints (17 Feb 2020), doi: https://doi.org/10.31219/osf.io/skznv.
Se están usando herramientas bioinformáticas para cribar (buscar) fármacos contra la proteasa principal de SARS-CoV-2 (en especial, los que se puedan reposicionar, que permiten acelerar los estudios clínicos). Por ejemplo, Anh-Tien Ton, Francesco Gentile, …, Artem Cherkasov, «Rapid Identification of Potential Inhibitors of SARS‐CoV‐2 Main Protease by Deep Docking of 1.3 Billion Compounds,» Molecular Informatics (11 Mar 2020), doi: https://doi.org/10.1002/minf.202000028; Zeshan Haider, Muhammad Muneeb Subhani, …, Adnan Khan Niazi, «In Silico Discovery of Novel Inhibitors Against Main Protease (Mpro) of SARS-CoV-2 Using Pharmacophore and Molecular Docking Based Virtual Screening from ZINC Database,» preprints (11 Mar 2020), doi: https://doi.org/10.20944/preprints202002.0431.v2; Sekhar Talluri, «Virtual Screening Based Prediction of Potential Drugs for COVID-19,» preprint (09 Mar 2020), doi: https://10.20944/preprints202002.0418.v2; Bowen Tang, Fengming He, …, Dong Xu, «AI-aided design of novel targeted covalent inhibitors against SARS-CoV-2,» bioRxiv (08 Mar 2020), doi: https://doi.org/10.1101/2020.03.03.972133; Muhammad Usman Mirza, Matheus Froeyen, «Structural Elucidation of SARS-CoV-2 Vital Proteins: Computational Methods Reveal Potential Drug Candidates Against Main Protease, Nsp12 RNA-dependent RNA Polymerase and Nsp13 Helicase,» preprint (05 Mar 2020), doi: https://doi.org/10.20944/preprints202003.0085.v1; Ren Kong, Guangbo Yang, …, Shan Chang, «COVID-19 Docking Server: An interactive server for docking small molecules, peptides and antibodies against potential targets of COVID-19,» arXiv:2003.00163 [q-bio.BM] (29 Feb 2020).
Por cierto, las simulaciones por dinámica molecular parecen indicar que los inhibidores de Mpro que son moléculas pequeñas parecen ser ineficaces, recomendándose moléculas más grandes. Más información en Maria Bzówka, Karolina Mitusińska, …, Artur Góra, «Molecular Dynamics Simulations Indicate the COVID-19 Mpro Is Not a Viable Target for Small-Molecule Inhibitors Design,» bioRxiv (02 Mar 2020), doi: https://doi.org/10.1101/2020.02.27.968008. Por fortuna, ya se han publicado múltiples estudios sobre fármacos inhibidores de la proteasa principal de SARS-CoV-2, por ejemplo, Zhenming Jin, Xiaoyu Du, …, Haitao Yang, «Structure of Mpro from COVID-19 virus and discovery of its inhibitors,» bioRxiv preprint (10 Mar 2020), doi: https://doi.org/10.1101/2020.02.26.964882; Xin Liu, Xiu-Jie Wang, «Potential inhibitors for 2019-nCoV coronavirus M protease from clinically approved medicines,» bioRxiv preprint (29 Jan 2020), doi: https://doi.org/10.1101/2020.01.29.924100; entre otros.
[PS 13 abr 2020] Se ha publicado en Nature el artículo de Jin et al. en bioRxiv: Zhenming Jin, Xiaoyu Du, …, Haitao Yang, «Structure of Mpro from COVID-19 virus and discovery of its inhibitors,» Nature 582: 289-293 (09 Apr 2020), doi: https://doi.org/10.1038/s41586-020-2223-y. [/PS]
[PS 24 abr 2020] Se ha publicado en Science el artículo de Linlin Zhang, Daizong Lin, …, Rolf Hilgenfeld, «Crystal structure of SARS-CoV-2 main protease provides a basis for design of improved α-ketoamide inhibitors,» Science 368: 409-412 (24 Apr 2020), doi: https://doi.org/10.1126/science.abb3405. [/PS]
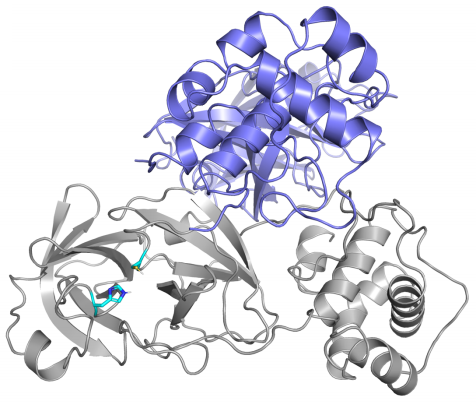
Esta es la primera reconstrucción por ordenador de la proteasa principal de SARS-CoV-2 que se publicó (según me consta); está basada en su homología con SARS-CoV. Aunque en la primera figura de esta pieza aparece el dímero (formado por dos copias de la proteína) que se ha cristalizado y aquí solo se presenta el monómero, se puede comprobar que son muy similares entre sí. Por fortuna, pues varios artículos posteriores se han basado en ella para estudiar el efecto de algunos fármacos inhibidores de su actividad.
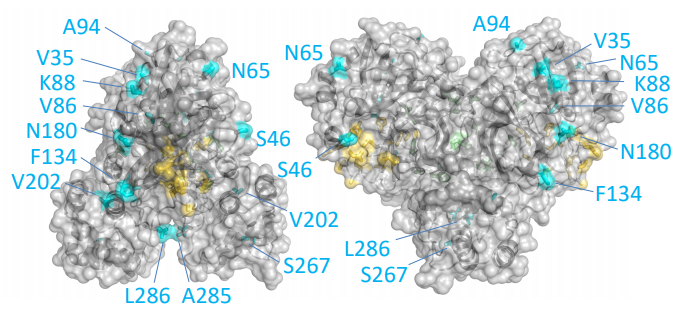
Esta reconstrucción de la proteasa principal (Mpro) del coronavirus SARS-CoV-2 muestra coloreados en celeste los residuos (aminoácidos) diferentes con dicha proteína de SARS-CoV. Solo se diferencian en 12 aminoácidos: V35L, S46A, N65S, V86L, K88R, A94S, F134H, N180K, V202L, S267A, A285T, y L286I; repito lo que ya dije más arriba, para quienes no conozcan la notación, por ejemplo, F134H (Phe134His) significa que SARS-CoV-2 tiene una F (Phe o fenilalanina) en la posición 134 de su secuencia de aminoácidos, donde SARS-CoV tiene una H (His o histidina), y así con todos los demás (tabla de aminoácidos). Puedes consultar las secuencias de referencia NP_828863.1 (SARS-CoV) y YP_009725301.1 (SARS-CoV-2).
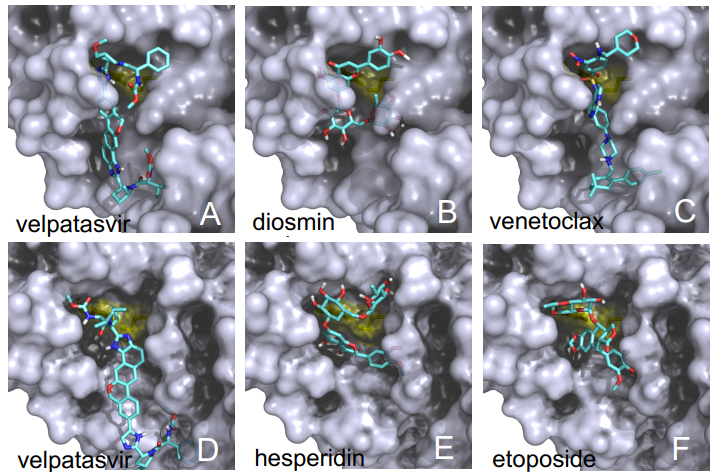
Las diferencias entre proteasa principal de SARS-CoV-2 y SARS-CoV son tan pequeñas que podemos confiar en la reconstrucción por homología de su estructura tridimensional. Gracias a ello podemos estudiar cómo interaccionan con esta proteasa diferentes antivirales, tanto los que atacan la cadena A (velpatasvir, diosmin y venetoclax) como los que atacan la cadena B (velpatasvir, hesperidin y etoposide). Por supuesto hay muchos más antivirales que inhiben esta proteasa, los de la figura son solo unos ejemplos representativos.
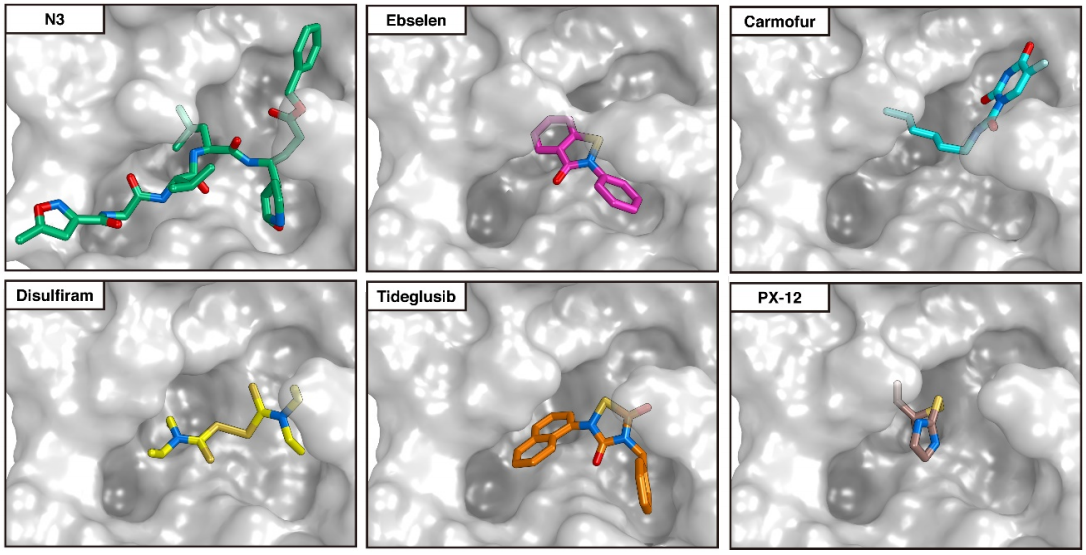
Esta figura muestra la predicción para la colocación (docking pose) de otros seis inhibidores de la proteasa principal en su sitio de acción. En concreto, N3, ebselen, carmofur, disulfiram, tideglusib y PX-12. Se han estudiado muchos más y se han realizado diferentes comparaciones entre ellos; por lo que parece son más eficaces las moléculas más grandes que parecen inhibir de forma más completa el sitio activo de la proteasa.
En resumen, se está trabajando mucho en el desarrollo de inhibidores de la proteasa principal del coronavirus SARS-CoV-2. Muchos se están probando en diferentes ensayos clínicos (alguno incluso usan varios en cócteles). En los próximos meses se publicarán los resultados (algunos serán prometedores, otros serán decepcionantes), pero auguro que antes de finales de año tendremos varias terapias exitosas ya ensayadas contra COVID-19. Las vacunas tardarán más en llegar, pero creo que son menos urgentes. La ciencia, como siempre, dando respuestas a velocidad de vértigo, aunque todo el mundo sueña con que lo hiciera a velocidad superlumínica, algo imposible. Confío en la ciencia. Soy optimista.


Excelente resumen
gracias muy buen articulo. Yo también confió absolutamente en la ciencia.
Saludos
Confío en la ciencia como conocimiento objetivo respetando otro tipo de conocimiento
Impresionantes las imágenes «tridimensionales» e impresionante el despliegue tecnológico y científico para afrontar una de las mayores crisis mundiales de las últimas décadas. Es increíble como una mutación que produce el acercamiento de 2 átomos de carbono apenas 1,5 A puede derivar (supuestamente) en un aumento importante de la capacidad de contagio (probablemente hubo anteriormente innumerables mutaciones antes de dar con la estructura más óptima). Con respecto a la última frase de Francis: «La ciencia, como siempre, dando respuestas a velocidad de vértigo… Confío en la ciencia. Soy optimista» Exactamente, casi nadie se acuerda de la ciencia en la vida cotidiana, sin embargo, cuando hay que tratar de comprender algún fenómeno «extraño» o «inusual» que irrumpe de repente en nuestras vidas cotidianas todo el mundo reclama a la ciencia una respuesta rápida y contundente. La ciencia nos sacará de esta crisis como ya nos ha sacado de otras muchas anteriormente principalmente evitando su aparición como consecuencia del avance de la medicina (antibióticos, vacunas, etc) si hubiéramos tenido antibióticos en la edad media hubiéramos evitado la muerte de la mitad de la población europea del momento debido a la peste y quizás también la de 50 millones de personas durante la mal llamada gripe española (creo que la neumonía que causaba era bacteriana). Para terminar quisiera hacer solo un par de apuntes: 1) Creo que es muy posible que el virus acabe siendo estacional como el virus de la gripe común y que una vez que tengamos vacunas y la población se inmunice tendremos la infección controlada 2) Lamentablemente España va a ser uno de los países más perjudicados debido a su dependencia del turismo y la hostelería. Hay que recordar también que la economía salva vidas (Alemania tiene más del doble de UCIs por habitante que España) y que la destrucción de la economía del país quizás genere más muertos que el propio virus (deterioro de la sanidad, empobrecimiento general, posible aumento del índice de suicidios, etc). Esperemos que la crisis no dure mucho y que las medidas adoptadas nos ayuden a paliar este «escenario catastrófico» que se avecina.
Conclusión: Esas personas con bata blanca que realizan «extraños experimentos» en laboratorios serán los encargados de «salvar el mundo» aunque nadie les rinda homenaje ni se hagan películas sobre ellos… Un saludo a todos los lectores de Francis y mucho ánimo a todos los que viven en estado de aislamiento.
Sigo habitualmente los interesantes posts de este blog. Soy un lector comùn con conocimiento promedio en estos temas, por eso, ya que se trata aquì de divulgaciòn,me surgen unas dudas de la lectura:
«El ARN de los coronavirus codifica poliproteínas que se escinden en varias proteínas responsables de su replicación en la célula huésped. Las proteasas que producen la escisión son dianas terapéuticas preferentes para los fármacos antivirales.»
No me queda claro:
– si las proteasas las genera el ARN del virus o son un agente defensivo generado por la cèlula huesped.
– si el ARN codifica pliproteìnas, estas se generaràn cuando el mismo es decodificado y replicado, entonces no entiendo còmo las proteìnas en las que se escinden dichas poliproteìnas pueden ser responsables de su replicaciòn.
Un saludo y perdone mi ignorancia.
Julio, en bacterias cada gen suele codificar una única proteína, sin embargo en muchos virus el ARN está muy empaquetado y un único gen puede codificar varias proteínas; entonces se dice que dicho gen codifica una poliproteína. Cuando los ribosomas de la célula huésped traducen el ARN del virus (que confunden con ARN mensajero propio) en proteínas producen estas poliproteínas.
Las primeras poliproteínas producidas incluyen una proteasa que es capaz de cortar (aunque de forma poco eficiente) las nuevas poliproteínas que se traducen; dichos cortes ocurren en ciertos lugares (llamados sitios de escisión); así las nuevas poliproteínas se rompen en proteínas individuales, siendo proteasas las primeras proteínas que aparecen tras la escición. Gracias a ello, en futuras traducciones, con proteasas ya disponibles la escisión de las poliproteínas en sus proteínas constituyentes es mucho más eficiente.
Para la replicación es fundamental que actúen las replicasas, que también están codificadas dentro de la poliproteínas. Estas replicasas no pueden actuar formando parte de la poliproteína (como ocurre con las proteasas); por tanto se requiere la acción de las proteasas para escindir las replicasas que se encuentran en las poliproteínas y permitir la replicación del virus. Por ello, si inhibes la acción de las proteasas, inhibes la replicación del virus.
Gracias por la información. Quiero preguntarte sobre la última figura con los 6 seis inhibidores de la proteasa (N3, ebselen, carmofur, disulfiram, tideglusib, PX-12); me podrías dar las referencias , por favor, o tú mismo hiciste la figura? De ser así, cuál software usas? Gracias de antemano.
Sacosta, como puedes leer en el nombre de la figura «https://francis.naukas.com/files/2020/03/D20200319-biorxiv-2020-02-26-964882-Docking-Poses-different-COVID-19-virus-Mpro-inhibitors.png» la fuente es el artículo «Structure of Mpro from COVID-19 virus and discovery of its inhibitors» https://www.biorxiv.org/content/10.1101/2020.02.26.964882v2
Muchas gracias por su excelente y clara explicación, señor Villatoro.Es una síntesis brillante.
Gracias por el post Francis, muy clarificador y con muy buenas referencias. Sólo un alcance, me parece que la enzima no contiene iones calcio, seguramente los átomos que se acercan son los carbonos alfa de los residuos involucrados.
Obviamente, David, craso error por mi parte, no entiendo por qué nadie se dio cuenta antes… obviamente son carbonos.
cual es su sitio de union especifico de la proteasa Mpro por favor