La infección de una célula hospedadora por el coronavirus SARS-CoV-2 requiere la fusión de sus membranas. Aún no se entiende bien cómo ocurre este proceso a nivel molecular. Un paso previo es la unión al receptor ACE2 que gracias a la criomicroscopia electrónica estamos empezando a entender. Se acaba de publicar en Nature el último avance en este línea, el estudio de los cambios conformacionales en la unión del trímero de proteínas S que forma la espícula a tres receptores ACE2. Se observa la aparición de una cavidad formada por los dominios S1 que expone al medio la parte superior de los dominios S2, donde se encuentra la región de heptámeros repetidos clave en la fusión de membranas. La conformación del complejo S1+ACE2 debilita la unión con S2 y permite que una proteasa escinda los dominios S1 y S2. La biología estructural del proceso es muy complicada pero poco a poco se van desvelando sus detalles más íntimos.
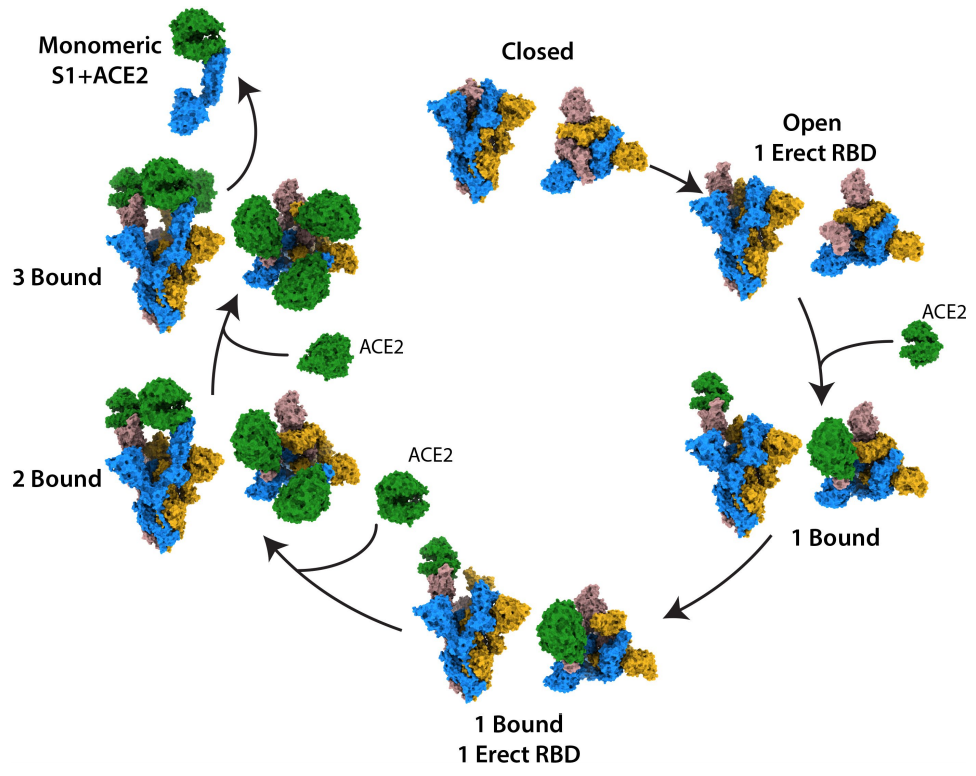
Esta figura ilustra un proceso complicado que ocurre rara vez (solo se ha observado en el 3 % de los complejos estudiados), la unión de una espícula a tres receptores ACE2 en la membrana celular. Se parte del estado cerrado (Closed) pasando a la apertura del sitio de unión al receptor de una de las proteínas del trimero (Open, 1 Erect RBD). Se une a un receptor ACE2 (1 Bound) y se abre otra de las proteínas del trímero (1 Bound, 1 Erect RBD). Se une un segundo receptor ACE2 (2 Bound), abriéndose la última proteína del trímero y unióndose a un tercer receptor (3 Bound). Finalmente, se observa la excisión de S1 y S2 dando lugar a monómeros del complejo S1+ACE2. Este proceso conduce a que queden expuestas regiones de heptámeros repetidos que permiten la fusión de las membranas (pero dicho proceso no se ilustra en esta figura). Aunque la unión más probable es la de la espícula a un solo receptor ACE2, los resultados apuntan a que la unión a dos receptores (solo algo menos probable) y la unión a tres receptores (muy poco probable) facilitan la infección.
El nuevo artículo es Donald J. Benton, Antoni G. Wrobel, …, Steven J. Gamblin, «Receptor binding and priming of the spike protein of SARS-CoV-2 for membrane fusion,» Nature (17 Sep 2020), doi: https://doi.org/10.1038/s41586-020-2772-0, continuación de su trabajo previo Antoni G. Wrobel, Donald J. Benton, …, Steven J. Gamblin, «SARS-CoV-2 and bat RaTG13 spike glycoprotein structures inform on virus evolution and furin-cleavage effects,» Nature Structural & Molecular Biology 27: 763-767 (09 Jul 2020), doi: https://doi.org/10.1038/s41594-020-0468-7.
Si necesitas más información para entender mejor esta pieza te recomiendo leer «El genoma del coronavirus chino 2019-nCov (ahora SARS-CoV-2)», LCMF, 25 ene 2020; «La estructura 3D de la glicoproteína espicular del coronavirus SARS-CoV-2», LCMF, 24 feb 2020; «La estructura 3D del receptor ACE2 que usa el coronavirus SARS-CoV-2 para infectar», LCMF, 07 mar 2020; «Cómo se une el coronavirus SARS-CoV-2 al receptor ACE2 de la célula huésped», LCMF, 31 mar 2020.
[PS 22 sep 2020] Se ha propuesto el uso de una versión trimérica del receptor ACE2 humano como antiviral contra SARS-CoV-2 en «A trimeric human angiotensin-converting enzyme 2 as an anti-SARS-CoV-2 agent in vitro,» bioRxiv preprint (18 Sep 2020), doi: https://doi.org/10.1101/2020.09.18.301952. [/PS]
La espícula está formada por tres proteínas S unidas formando un trímero; la proteína S está formada por dos dominios, llamados S1 y S2. El trímero tiene un tallo helicoidal central formado por los tres dominios S2 y una cabeza formada por los tres dominios S1. Cada dominio S1 consta a su vez de dos grandes subdominios, el dominio N-terminal (NTD) y el dominio de unión al receptor (RBD), entre los que se encuentra un subdominio intermedio más pequeño. El RBD tiene dos configuraciones llamadas abierta (open-RBD) y cerrada (closed-RBD); en la abierta, el receptor ACE2 del hospedador puede acceder a la parte superior del dominio S2, mientras que en la cerrada está inaccesible, impidiendo la unión. En estudios previos por criomicroscopia electrónica en los que se ha usado furina para inducir la transición de la configuración cerrada a la abierta se ha observada una conformación intermedia de RBD; así se cree que la furina puede «activar» el trímero para permitir la unión al receptor ACE2.

Mediante criomicroscopia electrónica se ha estudiado la unión entre la espícula del coronavirus SARS-CoV-2 y el receptor ACE2. Se han resuelto nueve estructuras distintas del trímero (tres proteínas S unidas que forma la espícula) y del complejo S-ACE2 tras aplicar un baño de furina durante ~60 segundos previo a la criogenización (véase la figura): el trímero cerrado (10.5 %), un estado intermedio del trímero con una de las proteínas semiabiertas debido a la interacción con la furina (4.1 %), un trímero con una proteína S abierta (15.6 %), un trímero con dos proteínas S abiertas (4.4 %), el complejo de ACE2 unido a una proteína abierta del trímero con las otros dos cerradas (28.1 %), el complejo de ACE2 unido a una proteína abierta del trímero con otra abierta en sentido horario y la otra cerrada (13.6 %), el complejo de ACE2 unido a una proteína abierta del trímero con otra abierta en sentido antihorario y la otra cerrada (7.4 %), el complejo de dos ACE2 unidas a sendas proteínas abiertas del trímero con la otra cerrada (13.6 %) y el complejo de tres ACE2 unidas a sendas proteínas abiertas del trímero (2.7 %).

La comparación entre el trímero abierto y el complejo con una ACE2 unida al trímero muestra que la unión rota el RBD de la S1 abierta alejando su centro de masas ~5.5 Å respecto al eje del trímero y al mismo tiempo aleja los tres NTD de las S2 del trímero ~1.5–3.0 Å; la unión a una o dos ACE2 adicionales no introduce cambios significativos en la conformación de la primera unión. Estos cambios de conformación estabilizan la unión de ACE2 en el complejo. Cuando se unen tres receptores ACE2 al complejo (figura de abajo) dejan descubierta (un-shield) la parte superior del tallo S2 a través de una especie de anillo; este anillo trimérico está formado de por dominios S1 unidos al tallo S2 solo a través de los pequeños subdominios intermedios. Entre el anillo y las tres ACE2 se forma una cavidad de ~50 Å de diámetro centrada en el eje del trímero que tiene ~65 Å de profundidad. Así quedan expuestos los aminoácidos 980-990 dentro de la región de heptámeros repetidos 1 (HR1) en la parte superior de S2, clave para la fusión de membranas entre el coronavirus y la célula hospedadora. El artículo discute en detalle los aminoácidos implicados en la formación de esta cavidad, así como sus enlaces químicos (los interesados pueden consultar el artículo); por desgracia, la conformación helicoidal detallada de las regiones HR1 no se puede determinar por criomicroscopia electrónica.
Los resultados apuntan a que la probabilidad de fusión entre membranas está favorecida por el número de receptores ACE2 unidos a la proteína espicular. Así la infección sería más probable en células que expresen un gran número de receptores ACE2. Hay muchos detalles moleculares de la fusión de las membranas que aún se ignoran. La relevancia clínica potencial de estos resultados no es lo que más me interesa, lo siento; pero me fascina todo lo que estamos aprendiendo gracias a la criomicroscopia electrónica. La belleza es una parte fundamental de la ciencia y la biología estructural es una de las ramas más bellas de la biología.


Sería interesante saber si esa es el caso en el murciélago o el pangolín, supongo.
En su artículo previo citado (https://doi.org/10.1038/s41594-020-0468-7) los autores estudiaron la proteína espicular del coronavirus de murciélago RaTG13, el más próximo a SARS-CoV-2 entre los conocidos (en concreto su unión hasta con dos ACE2). No se ha estudiado la del pangolín, ni creo que se estudie.
En realidad hay que matizar que el RBD de la proteína espicular de S1 en el actual coronavirus es muy parecido a la secuencia homóloga de un pangolin decomisado en Guangdong en marzo de 2019, incluyendo los cinco aminoácidos críticos para la óptima fijación al receptor. Posiblemente su gran afinidad por el ACE2 humano se debe a estas características. El resto del genoma sí guarda más parecido con RaTG13. Podríamos decir que S1 en el Sars CoV 2 es como el de RaTG13 con un RBD de pangolin insertado en medio. Este es otro detalle sorprendente de este virus pandémico que no resulta fácil de explicar.
La evolución en acción, amigo, la evolución. Se explica fácilmente, Paco, ya se conocen muchas inserciones y deleciones en las diferentes cepas secuenciadas respecto al genoma de referencia (el primero en ser secuenciado en enero); en los primeros hospedadores ocurrió una inserción como la que indicas que favoreció dicha afinidad. ¿Qué papel tuvo el coronavirus de pangolín? No se sabe, pero es muy probable que ninguno; así funciona la evolución, como sabe cualquiera que la ha estudiado.
Muchas gracias por tu respuesta a mi comentario. Aprovecho la ocasión para hacerte una petición y ampliar un poco el asunto de los coronavirus de pangolin en el orígen de la proteína S del Sars CoV 2.
1. Te agradecería más información sobre tu aseveración de que «ya se conocen muchas inserciones y deleciones en las diferentes cepas secuenciadas respecto al genoma de referencia (el primero en ser secuenciado en enero); en los primeros hospedadores ocurrió una inserción como la que indicas que favoreció dicha afinidad». Me gustaría disponer de acceso a la base de datos y/o bibliografía manejda.
2. En los coronavirus de pangolin estudiados en 2019 y anotados como pangolin-CoV-2020 la identidad de su RBD con respecto a la del virus pandémico actual es muy elevada, sugiriendo que aquél puede estar capacitado para fijarse directamente al receptor ACE2 humano. La procedencia de los pangolines no es bien conocida, pero podrían haber sido capturados en zonas de Malasia y transportados hasta China donde son decomisados en Guangdong. En todo ese viaje en cautividad pueden haber estado en estrecho contacto con murciélagos y otros animales antes de llegar a diferentes mercados de los paises del entorno. Es posible que ello haya favorecido eventos de recombinación que pueden haber propiciado el fenómeno de inserción que estamos comentado. En este sentido es notable que en un estudio amplio de más de 300 pangolines capturados entre 2009 y 2019 en la península malaya, en ningún caso se haya detectado por RT-PCR que fueran portadores de coronavirus. Esto recuerda el caso de las civetas en relación con el brote de SARS de 2002-2003, donde se detectó el virus en los animales en mercados y restaurantes pero no en ninguna de las múltiples granjas estudiadas.
En resúmen, creo que el papel de ciertos pangolines en el orígen del virus pandémico actual y particularmente en la constitución de la proteína S de éste, optimizando un RBD de alta afinidad por la ACE2 humana, no puede ser completamente descartado.
Paco, (1) si te interesa el tema suscríbete a la web de GISAID (https://www.gisaid.org/); ahora mismo almacena unos 112 mil genomas de SARS-CoV-2; además podrás acceder a los genomas completos así como a múltiples artículos/comentarios que los discuten. Si no te interesa tanto, pero quieres leer algunos artículos busca artículos que discutan las deleciones observadas en https://www.biorxiv.org/ (yo habré leído unos diez). Y (2) obviamente, que el pangolín sea intermediario entre murciélagos y humanos no se puede descartar de forma rotunda.
Bueno, pues pregunto, acojnado: es posible usar una tecnologia CRIPSR-Cas9 para «editar» ése RBD de pangolín insertado en el «esqueleto» del genoma del RaTG13?.
Anonimo, no se puede usar CRISPR-Cas9, que edita ADN de doble cadena, pero en el año 2019 se desarrolló una técnica CRISPR diferente para la edición de ARN. Existen otras técnicas de ingeniería genética mucho más antiguas que permiten cortar un trozo del ARN de un coronavirus de pangolín y ponerlo en el ARN de un coronavirus de murciélago; ahora bien, nadie sabe qué trozo se debe cortar para lograr un objetivo concreto, el que sea, pues nuestro conocimiento sobre los ARN de los coronavirus es insuficiente (lo era en 2019 y sigue siéndolo en 2020).
Es encomiable tu interés por los entresijos ultraprofundos de la mecánica por la que el actual coronavirus se une a su receptor canónico ACE2, pero debo decir que, aunque muy interesado en toda esta problemática, no llego a tu nivel de obsesión por los detalles.Parece razonable pensar que, puesto que la proteína S1 es trimérica, ha de exponer tres RBD para su potencial unión al receptor. Que coincida una molécula de ACE2 para cada uno de los RBD de S1 se antoja difícil. De ahí que en este estudio, que dicho entre paréntesis, me supera, sea más probable que una proteína S se fije a un receptor ACE2, que a dos o a tres. Pero la idea de que la fijación triple conduzca necesariamente a una infectividad mayor no tiene porqué correlacionarse con los hallazgos clínicos. Un planteamiento alternativo es que el virus tiene más asegurada su fijación a la célula si tiene tres opciones que si sólo tiene una, aunque con una le baste y le sobre para infectar a la mayoría de tejidos que expresen ACE2. Otra cuestión es el tema de la furina. Este virus tiene una secuencia PRRA, justo antes de la R por donde se produce el clivage usual en otros coronavirus, que le faculta para disponer de un amplio espectro de células en las que entrar una vez la secuencia es reconocida por la ubicua protesasa furina. No se dónde he leído que eliminando esa secuencia, el virus se comporta como el del SARS, y que en cambio su presencia le faculta para un organotropismo múltiple y, por ende, mayor daño multiorgánico. Como ves, a mi me interesa más la aplicación clínica del estudio. No obstante, la ciencia básica siempre es importante y alguna vez respaldará desarrollos prácticos de indudable interés. Un saludo.
Magnífico nivel de detalle. Admiro y comparto la pasión de Francis por la naturaleza de los procesos ya que derivan en entendimiento y aplicación clínica.
Gracias por el articulo Francisco!
Increíble la «maquinaria natural»…..
La Naturaleza es compleja y bella hasta el extremo.