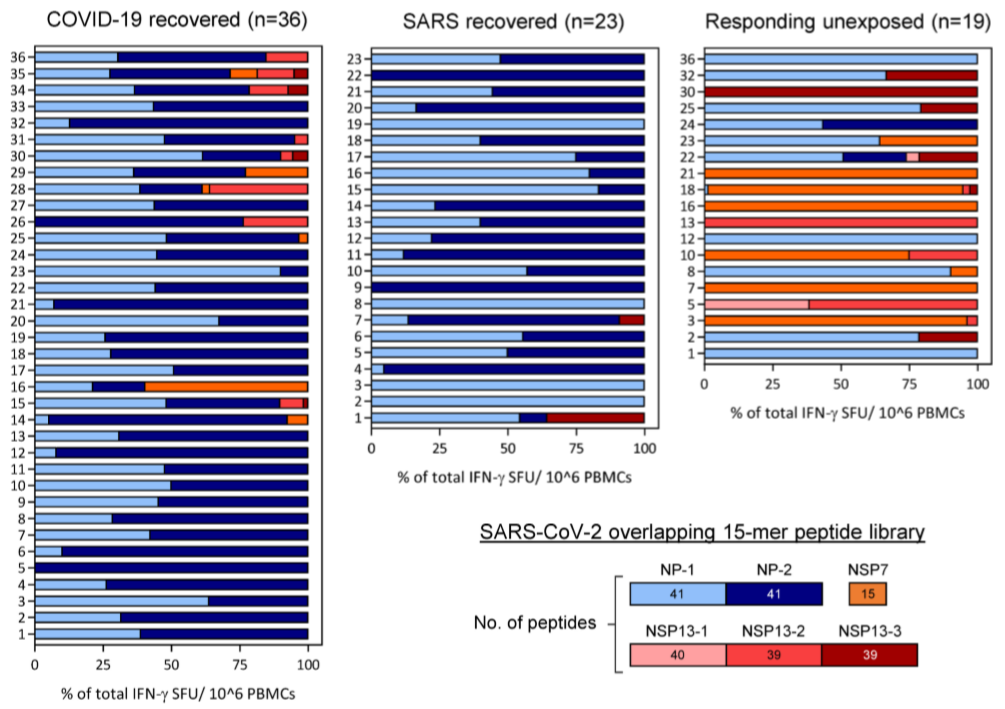
Los estudios de seroprevalencia no tienen en cuenta la inmunidad celular (la memoria inmune de los linfocitos T CD4+ y CD8+); por ello podrían subestimar el nivel de inmunidad a la COVID-19 entre la población. Por desgracia, como ocurre con la RT-PCR, las pruebas de respuesta inmune de linfocitos T son costosas y lentas usando citometría de flujo. Se ha publicado en Nature que 23 pacientes que superaron el SARS hace 17 años y 36 que han superado la COVID-19 tienen linfocitos T que reaccionan ante algunos epítopos del coronavirus SARS-CoV-2. Esta posible inmunidad cruzada entre sarbecovirus podría ser esperada, pero, además, en 19 personas de 37 no expuestas ni al SARS ni a la COVID-19 también se observa reactividad en sus linfocitos T ante estos epítopos; así se sugiere que podría haber una inmunidad cruzada entre betacoronavirus (como los que causan entre el 10 % y el 15 % de los resfriados).
Muy buenas noticias, pero hay que tomarlas con una pizca de sal. Por un lado, el número de pacientes del estudio es muy bajo, n = 96, lo que implica una estadística pobre; se requieren estudios mucho más extensos que limiten los posibles sesgos de confirmación en el análisis. Por otro lado, se ha estudiado la reactividad de los linfocitos T in vitro con epítopos de la proteína estructural N (epítopos NP-1 y NP-2) y de dos proteínas no estructurales de la poliproteína ORF1, en concreto NSP7 y NSP13 (epítopos NSP13-1, NSP13-2 y NSP13-3); todas ellas están bien conservadas entre los betacoronavirus, siendo muy parecidas entre los sarbecovirus. Pero, además, se encuentran dentro de la membrana del coronavirus, lo que podría conllevar que la reactividad in vivo de los linfocitos T sea mucho menor que in vitro.
Se requieren futuros estudios que consideren otros epítopos, en especial, de proteínas expuestas fuera de la membrana del coronavirus (como la proteína espicular S y las proteínas M y E); y estudios específicos dirigidos a verificar la reactividad cruzada entre betacoronavirus (mucho más sorprendente que entre sarbecovirus). Pero no quiero olvidar cierto optimismo; estos resultados podrían implicar que el desarrollo de vacunas que induzcan una respuesta inmune en los linfocitos T contra la COVID-19 podría ser más fácil de lo esperado. El artículo es Nina Le Bert, Anthony T. Tan, …, Antonio Bertoletti, «SARS-CoV-2-specific T cell immunity in cases of COVID-19 and SARS, and uninfected controls,» Nature (15 Jul 2020), doi: https://doi.org/10.1038/s41586-020-2550-z, bioRxiv preprint 115832 (27 May 2020), doi: https://doi.org/10.1101/2020.05.26.115832; recomiendo también Derek Lowe, «New Data on T Cells and the Coronavirus,» Blog, Science Translational Medicine, 15 Jul 2020 [web].
Sobre inmunidad celular a la COVID-19 recomiendo leer a Daniel M. Altmann, Rosemary J. Boyton, «SARS-CoV-2 T cell immunity: Specificity, function, durability, and role in protection,» Science Immunology 5: eabd6160 (17 Jul 2020), doi: https://doi.org/10.1126/sciimmunol.abd6160; Alba Grifoni, Daniela Weiskopf, …, Alessandro Sette, «Targets of T Cell Responses to SARS-CoV-2 Coronavirus in Humans with COVID-19 Disease and Unexposed Individuals,» Cell 181: P1489-1501.E15 (14 May 2020), doi: https://doi.org/10.1016/j.cell.2020.05.015; Alan Sariol, Stanley Perlman, «Lessons for COVID-19 immunity from other coronavirus infections,» Inmunity (14 Jul 2020), doi: https://doi.org/10.1016/j.immuni.2020.07.005. Además de Takuya Sekine et al. (Karolinska COVID-19 Study Group), «Robust T cell immunity in convalescent individuals with asymptomatic or mild COVID-19,» bioRxiv preprint 174888 (29 Jun 2020), doi: https://doi.org/10.1101/2020.06.29.174888;
En español recomiendo leer a Esther Samper, «¿Podría la exposición a otros coronavirus protegernos frente al virus SARS-CoV-2?», elDiario.es, 16 jul 2020; Jesús Méndez, «Defensas contra la COVID-19 en sangre de 2018: la potente hipótesis de la inmunidad cruzada», Agencia SINC, 23 may 2020; Juan José Castillo, «En los linfocitos T podría estar la clave contra el COVID-19», Yahoo!, 15 jul 2020.
[PS 21 jul 2020] Por cierto, un epítopo, también llamado determinante antigénico, es la pequeña secuencia de aminoácidos (aa) a la que se unen los anticuerpos y los linfocitos. En el artículo de Nature se ha dividido la proteína NP (419 aa) en dos bloques NP-1 (1-215) y NP-2 (206-419), y, a su vez, cada uno de ellos en 41 epítopos de 15 aa solapados en 10 (es decir, 1-15, 6-20, 11-25, …, 201‐215, 206-220, …, 406-419); la proteína NSP7 (83 aa) en 15 epítopos (1-15, 6-20, …, 69-83); y la proteína NSP13 (601 aa) en tres bloques NSP13-1 (1-210), NSP13-2 (201-405) y NSP13-3 (396-601), divididos en 40, 39 y 39, resp., epítopos de 15 aa solapados. Muchos epítopos de SARS-CoV y SARS-CoV-2 se diferencian en ninguno, uno o dos aminoácidos. Todos los detalles en la información suplementaria [PDF].
También recomiendo el artículo de Daniela Weiskopf, Katharina S. Schmitz, …, Rory D. de Vries, «Phenotype and kinetics of SARS-CoV-2–specific T cells in COVID-19 patients with acute respiratory distress syndrome,» Science Immunology 5: eabd2071 (26 Jun 2020), doi: https://doi.org/10.1126/sciimmunol.abd2071, bioRxiv preprint 20062349, doi: https://doi.org/10.1101/2020.04.11.20062349. [/PS]
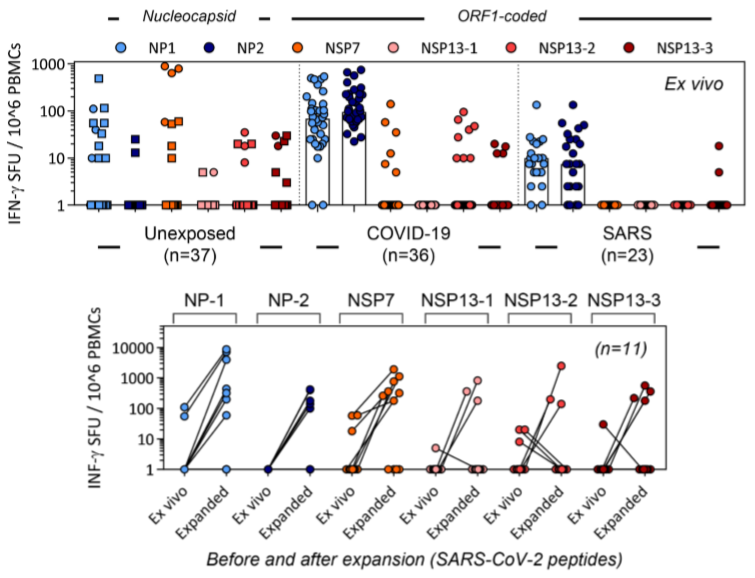
Esta figura muestra la reactividad de los linfocitos T a los seis epítopos estudiados; nota que se usa una escala logarítmica, así la respuesta de quienes han superado la COVID-19 es unas diez veces mayor a los epítopos NP1 y NP2 que para quienes han superado el SARS; además, estos últimos no presentan reactividad a los otros epítopos (NSP7, NSP13-1, NSP13-2 y NSP13-3). En cuanto a los no expuestos se observa mucho menos reactividad, aunque repartida entre todos los epítopos; la razón debe ser que fueron expuestos a resfriados provocados por betacoronavirus humanos como HCoV-OC43 y HCoV-HKU1. Con los datos en la mano, confiar en que uno está inmunizado contra la COVID-19 porque ha pasado muchos resfriados en los últimos lustros es muy arriesgado. En mi opinión hay que ser muy escéptico con la posible inmunidad cruzada entre betacoronavirus.
El resultado más relevante del nuevo trabajo es que la inmunidad cruzada entre los sarbecovirus (SARS-CoV-1 y SARS-CoV-2) implica que la inmunidad celular gracias a los linfocitos T en las infecciones por sarbecovirus tiene mucha mayor duración (al menos 17 años para el SARS) que la inmunidad humoral gracias a los anticuerpos (que se estima entre 2 y 3 años para el SARS). Por la gran similitud filogenética entre ambos sarbecovirus, se puede esperar una inmunidad celular para la COVID-19 con una duración similar. Un gran noticia para el desarrollo de vacunas que induzcan este tipo de respuesta inmune, como las vacunas de CanSino, Moderna y Oxford (the CanSino vaccine group, «Safety, tolerability, and immunogenicity of a recombinant adenovirus type-5 vectored COVID-19 vaccine: a dose-escalation, open-label, non-randomised, first-in-human trial,» The Lancet 395: P1845-1854 (22 May 2020), doi: https://doi.org/10.1016/S0140-6736(20)31208-3; the mRNA-1273 Study Group (Moderna), «An mRNA Vaccine against SARS-CoV-2 — Preliminary Report,» NEJM (14 Jul 2020), doi: https://doi.org/10.1056/NEJMoa2022483; the Oxford COVID Vaccine Trial Group, «Safety and immunogenicity of the ChAdOx1 nCoV-19 vaccine against SARS-CoV-2: a preliminary report of a phase 1/2, single-blind, randomised controlled trial,» The Lancet (20 Jul 2020), doi: https://doi.org/10.1016/S0140-6736(20)31604-4 [PDF]).

Por cierto, cuando se divulga este artículo de Nature solo se suele mostrar esta figura, que simplifica hasta el extremo los resultados. Los porcentajes parecen fáciles de entender para todo el mundo, pero ofrecen una información agregada que puede confundir a muchos. Sobre todo puede hacer creer que la inmunidad cruzada observada entre betacoronovirus en 19 pacientes es mayor de lo que en realidad es; compara esta figura con la anterior, donde se cuantifica la reactividad.
Me gustaría enfatizar que los epítopos de las proteínas usados en este estudio no se encuentran en la superficie del membrana del coronavirus. Tanto la fosfoproteína de la nucleocápside N (que es responsable de compactar el ARN del coronavirus en su interior), como la replicasa NSP7 y la helicasa de ARN NSP13 (que también interviene en la replicación), véase LCMF, 25 ene 2020, no pueden inducir la reactividad de los linfocitos T en las primeras fases del contagio. Se requiere que el coronavirus haya infectado una célula, se haya replicado (se alcanzan hasta cien mil copias) y se destruya dicha célula para que los viriones salgan al medio exterior para infectar otras células; en ese momento, cierta cantidad de proteínas N, NSP7 y NSP13 que quedaron en el citoplasma de la célula hospedadora, se exponen a la detección por parte de los linfocitos T tras la apoptosis de dicha célula.

Lo ideal habría sido usar epítopos de proteínas que se encuentran en la superficie del coronavirus (como la S o la M en esta figura de un manuscrito en bioRxiv del Karolinska COVID-19 Study Group); incluso sobre la proteína E, la que la OMS considera imprescindible para una detección firme del coronavirus mediante RT-PCR. La verdad, no sé por qué no se han usado epítopos de dichas proteínas en este estudio (y por qué los revisores no se lo han exigido a los autores); creo que futuros estudios en esta línea son imprescindibles antes de extraer conclusiones apresuradas sobre este artículo publicado en Nature.
En resumen, un resultado fascinante y sorprendente, aunque nunca hay que olvidar cierta dosis de escepticismo. Todos estamos deseando oír buenas noticias, pero debemos ser muy cautos; nuestros deseos nunca deben estar por encima de lo que en rigor muestran los resultados obtenidos. A pesar de ello, me gusta ser optimista, confío en que podremos lidiar con el fuerte rebrote esperado para octubre y que lo superaremos llegando en buenas condiciones hasta la primavera de 2021; entonces empezaremos a disfrutar de los grandes avances en tratamientos y vacunas que se están logrando durante este año. La ciencia sigue avanzando a paso firme y seguro.


Francis revisa esta noticia que se ve intersante sobre un posible tratamiento. http://www.pharmatimes.com/news/synairgens_sng001_shows_strong_promise_in_covid-19_trial_1345119
Hola. buena divulgacion…
Unas preguntas
1. Hablas de las diferentes NSP, cito textualmente lo que escribes:
«En el artículo de Nature se ha dividido la proteína NP (419 aa) en dos bloques NP-1 (1-215) y NP-2 (206-419), y, a su vez, cada uno de ellos en 41 epítopos de 15 aa solapados en 10 (es decir, 1-15, 6-20, 11-25, …, 201‐215, 206-220, …, 406-419); la proteína NSP7 (83 aa) en 15 epítopos (1-15, 6-20, …, 69-83); y la proteína NSP13 (601 aa) en tres bloques NSP13-1 (1-210), NSP13-2 (201-405) y NSP13-3 (396-601), divididos en 40, 39 y 39, resp., epítopos de 15 aa solapados. Muchos epítopos de SARS-CoV y SARS-CoV-2 se diferencian en ninguno, uno o dos aminoácidos.»
Creo recordar un video de The Epoch Times, un lobby dem presion contra el partido Comunista Chino en la cul salía una supuesta cientifica, Judy Mitckovitz creo que se llama y que decía que la NSP7 es 100 por cien id´´entica a la del virus …HIV.
Bueno, esto me parece pseudociencia, pero n9o tengo mas remedio que decirlo: esa cientifica fue detenida en 2011 por un fraude en los datos sanitarios.
En fin, que si ya de por si la ciencia es complicada, estos pseudocientificos complican mas la Verdad…
Nemo, te refieres a Judy Mikovits; recomiendo leer sobre su fraude científico en 2009 en Stuart J.D. Neil, Edward M. Campbell, «Fake Science: XMRV, COVID-19, and the Toxic Legacy of Dr. Judy Mikovits,» AIDS Research and Human Retroviruses 36: 545-549 (15 May 2020), doi: http://doi.org/10.1089/aid.2020.0095.
En cualquier caso, puedes comprobar fácilmente que el VIH no tiene ninguna proteína llamada NSP7 o que sea similar a la NSP7 del SARS-CoV-2. Compruébalo tú mismo: NSP7 (https://www.ncbi.nlm.nih.gov/protein/1802476811), HIV 1 (https://www.ncbi.nlm.nih.gov/nuccore/NC_001802.1) y HIV 2 (https://www.ncbi.nlm.nih.gov/nuccore/9628880).